서언
재료 및 방법
실험재료
시료의 추출
유리당 함량 분석
결과 및 고찰
무 유전자원의 외형적 특성
총 유리당 함량
유전형에 따른 유리당 함량 분석
자원의 구분에 따른 유리당 함량 분석
적요
서언
무(Raphanus sativus L.)는 십자화과(Brassicaceae)에 속하는 뿌리채소 작물로 전세계적으로 재배되어 이용되고 있다. 일본, 한국, 그리고 중국을 포함한 동아시아권과 유럽을 중심으로 잎과 뿌리 부분을 채취하여 다양한 요리 재료로 활용하고 있으며, 열대 아시아에서는 꼬투리 부분을 식용으로 섭취하기도 하며, 일부 품종의 경우는 사료 및 유지 작물(convar. oleifer Alef.)로 재배가 이루어지고 있다(Hashida et al., 2013). 최근에는 무에 함유된 비타민, 미네랄, 섬유질, 그리고 항암 및 항산화 물질로 알려진 글루코시놀레이트의 풍부한 함유량이 알려지면서 기능성 채소 작물로 각광을 받고 있다(Ko et al., 2017; Yi et al., 2016). 무는 잎과 뿌리의 모양, 색, 크기 등 형태적으로 다양한 특징을 가지며, 조직경도, 개화시기, 영양성분 등의 생리적 차이가 다양하여 품질이 우수한 무의 개발 및 생산을 위해서는 야생종과 재래종 등을 포함한 다양한 유전자원의 확보가 매우 중요하다(Ellstrand and Marshall, 1985). 최근에는 대용량 염기서열 분석 기술(NGS)이 발달하면서 다양한 무 유전자원의 표준유전체 해독 연구가 수행되었다(Jeong et al., 2016; Kitashiba et al., 2014; Mitsui et al., 2015; Moghe et al., 2014; Xiaohui et al., 2015). 그리고 우수 계통에 대한 유전체 비교 분석과 이를 통한 유용 형질 관련 유전자의 탐색 및 분자 표지 개발연구(Kim et al., 2016; Mun et al., 2015)가 활발히 이루어지면서 체계적이고 종합적인 무 유전자원의 연구체계 구축의 필요성이 대두되고 있는 실정이다.
무의 단맛을 결정짓는 요소 중에 하나인 유리당의 분포는 무의 품질지표로서 중요한 의미를 지니고 있다(Lee et al., 2006). 식물의 중요한 탄소공급원인 유리당은 조직의 단맛에 영향을 미칠 뿐 아니라 다양한 환경적 변화에 대응하는 식물의 방어기작을 조절하는 것으로 알려져 있다(Dobrenel et al., 2013; Koch K., 2004). 무의 유리당은 주로 포도당, 과당, 자당이며, 때로는 이당류인 엿당을 포함한다(Hara et al., 2011; Kwak et al., 2017). 포도당(glucose)은 식물의 에너지원으로 사용되는 탄수화물 대사의 중요한 화합물로서 식물의 신진대사 및 에너지 생성을 위해 중요한 역할을 담당하고 있다(Koster et al., 1988). 자당(sucrose)과 과당(fructose)은 건조 및 염 스트레스와 같은 식물의 외부 환경 변화에 대응하는 역할을 하는 것으로 알려져 있다(Pilon-Smits et al., 1995). 2개의 포도당이 탈수합성(Dehydration synthesis)으로 결합한 이당류인 엿당(maltose)은 식물의 전분 생합성에서 중요한 역할을 담당하며, 식물의 circadian과 관련된 일장 그리고 고온 스트레스에 관여하는 것으로도 알려져 있다(Lu and Sharkey, 2006). 무의 유리당 관련 연구는 몇몇 품종에 대한 유리당 함량의 차이에 관한 보고(Hara et al., 2011)가 있으나, 광범위한 유전자원을 대상으로 유리당 함량을 분석하거나 유리당 생합성에 영향을 미치는 요소를 규명한 연구는 아직까지 보고되지 않고 있다. 본 연구에서는, 다양한 형질을 가진 무의 유전자원을 수집하여 무에서 분포하는 것으로 알려진 포도당, 자당, 과당, 그리고 엿당의 네 가지 유리당 함량을 분석함으로써 고품질 무 유전자원을 선발하고, 유리당 합성에 관여하는 메커니즘을 규명하는데 유용한 자료로 활용할 예정이다.
재료 및 방법
실험재료
본 연구의 재료로 사용한 무는 한국, 독일, 그리고 일본에서 보유하고 있는 유전자원을 수집하여 사용하였다. 국립농업과학원 농업유전자원센터((Rural Development Administration (RDA), http://genebank.rda.go.kr)에서 분양 받은 45 계통(IT-), 독일의 External Branch North of the Department Genebank, IPK, Oil Plants and Fodder Crops in Malchow (Institute of Plant Genetics and Crop Plant Research (IPK), www.genesys-pgr.org/)에서 분양받은 5계통(CR-)과 Leibniz Institute of Plant Genetics and Crop Plant Research (http://www.ipk-gatersleben.de/en/genebank/)에서 분양 받은 24계통(RA-), 그리고 일본의 농업생물자원 Genebank (National Agriculture and Food Research Organization (NARO), www. gene.affrc.go.jp)로부터 분양 받은 8계통(JP-)을 포함한 총 82 계통의 유전자원(Table 1)을 사용하여 유리당 함량을 분석하였다. 유전자원은 모두 1-2번의 뇌수분을 통한 자가수분을 실시하여 고정화한 계통들을 사용하였다.
Table 1. Information about 82 accessions of R. sativus
| Accession | Name | species | Origin | Type |
| CR1787 | Slobolt | Raphanus sativus convar. oleifer
(Stokes) Alef. | United Kingdom | Improved
cultivar |
| CR2970 | Tetra Poznanska | Raphanus sativus convar. oleifer
(Stokes) Alef. | Poland | Improved
cultivar |
| CR3472 | nabo | Raphanus sativus convar. oleifer
(Stokes) Alef. | Cuba | Traditional
cultivar |
| CR499 | Unknown | Raphanus sativus convar. oleifer
(Stokes) Alef. | Pakistan | Traditional
cultivar |
| CR504 | Unknown | Raphanus sativus convar. oleifer
(Stokes) Alef. | Canada | Traditional
cultivar |
| IT 102547 | Nursery No. 3007 | Raphanus raphanistrum | USA | Wild |
| IT110840 | Jinju-Daephyeong | Raphanus sativus var. sativus | South Korea | Improved
cultivar |
| IT 110843 | Gaeseong Altari Mu | Raphanus sativus var. sativus | South Korea | Improved
cultivar |
| IT 113530 | Chokseong Daehyeong
Bom Mu(214S) | Raphanus sativus var. sativus | South Korea | Improved
cultivar |
| IT 119228 | PI138636 | Raphanus sativus var. sativus | Iran | Wild
relatives |
| IT 119235 | PI138644 | Raphanus sativus var. sativus | Iran | Wild
relatives |
| IT 119238 | PI138647 | Raphanus sativus var. sativus | Iran | Wild
relatives |
| IT 119252 | Muli | Raphanus sativus var. sativus | India | Wild
relatives |
| IT 119279 | PI181802 | Raphanus sativus var. sativus | Syria | Wild
relatives |
| IT 119281 | Hamsi | Raphanus sativus var. sativus | Syria | Wild
relatives |
| IT 119282 | PI181805 | Raphanus sativus var. sativus | Lebanon | Wild
relatives |
| IT 119287 | PI211744 | Raphanus sativus var. sativus | Afghanistan | Wild
relatives |
| IT 119294 | Rozovo-krasny Belym
Konchikom | Raphanus sativus var. sativus | Russia | Wild
relatives |
| IT 119299 | Dungansky | Raphanus sativus var. sativus | Russia | Wild
relatives |
| IT 119304 | Minowase | Raphanus sativus var. sativus | Russia | Wild
relatives |
| IT 119318 | PI269597 | Raphanus sativus var. sativus | Pakistan | Wild
relatives |
| IT 119319 | Crvena Bela | Raphanus sativus var. sativus | Yugoslavia | Wild
relatives |
| IT 136490 | Pakhribas Local | Raphanus sativus var. sativus | Nepal | Traditional
cultivar |
| IT 138209 | BOL39 | Raphanus sativus var. sativus | Unknown | Wild
relatives |
| IT 180726 | Unknown | Raphanus sativus var. sativus | Egypt | Wild
relatives |
| IT 186722 | Intermediate parent
Nong 3 ho | Raphanus sativus var. sativus | Japan | Wild
relatives |
| IT 188101 | Unknown | Raphanus sativus var. sativus | China | Wild
relatives |
| IT 203302 | Local-Bukhara | Raphanus sativus var. sativus | Uzbekistan | Traditional
cultivar |
| IT 209972 | Hanoi | Raphanus sativus var. sativus | Vietnam | Unknown |
| IT 215053 | Danji Mu | Raphanus sativus var. sativus | South Korea | Traditional
cultivar |
| IT 216713 | Wild relatives
(Jejuseogwipo
-2003-21) | Raphanus sativus var. raphanistroides | Korea, South | Wild
relatives |
| IT 250744 | MMR-JYH-2010-63 | Raphanus sativus var. sativus | Myanmar | Traditional
cultivar |
| IT 250748 | KO18 | Raphanus sativus var. sativus | Turkey | Traditional
cultivar |
| IT 250766 | Sango | Raphanus sativus var. sativus | Italy | Traditional
cultivar |
| IT 250769 | CHN-CHOMC-2002-82 | Raphanus sativus var. sativus | China | Improved
cultivar |
| IT 250770 | THA-SSJ-2002-1 | Raphanus sativus var. sativus | Thailand | Improved
cultivar |
| IT 250780 | Redlong | Raphanus sativus var. sativus | India | Improved
cultivar |
| IT 250793 | Kohlschwarzer
Plattrunder | Raphanus sativus var. niger | Unknown | Unknown |
| IT 261002 | Wild relatives (Busan
Gijang-2003-39) | Raphanus sativus var. raphanistroides | South Korea | Wild
relatives |
| IT 261947 | Jingxuan nan pan
zhou meng bo | Raphanus sativus var. sativus | China | Improved
cultivar |
| IT 261997 | SAXA | Raphanus sativus var. sativus | Chile | Improved
cultivar |
| IT 261999 | A FORCER GOLO | Raphanus sativus var. sativus | France | Improved
cultivar |
| IT 262007 | Yeonjihong | Raphanus sativus var. sativus | China | Improved
cultivar |
| IT 262017 | CHN-YAAS-2010-93 | Raphanus sativus var. sativus | China | Improved
cultivar |
| IT 263949 | Wild relatives
(Namjeju-KHC-1999-44) | Raphanus sativus var. raphanistroides | Korea, South | Wild relatives |
| IT 276363 | UZB-HHS-2008-38 | Raphanus sativus var. sativus | Kyrgyzstan | Traditional
cultivar |
| IT 276488 | CHN-YAAS-2010-97 | Raphanus sativus var. sativus | China | Improved
cultivar |
| IT 277626 | Guanun-AN 6-4 | Raphanus sativus var. sativus | Korea, South | Improved
cultivar |
| IT 278270 | CHN-GHC-2003-2 | Raphanus sativus var. sativus | China | Traditional
cultivar |
| IT 291148 | National rond
about blan | Raphanus sativus var. sativus | Algeria | Unknown |
| JP26959 | HOKKAI CHOZOU 1 | Raphanus sativus var.
longipinnatus L.
H. Bailey (Daikon Group) | Japan | Unknown |
| JP27208 | AKIDOMARI DAIKON | Raphanus sativus var.
longipinnatus L.
H. Bailey (Daikon Group) | Japan | Unknown |
| JP27211 | SHIN RISOU | Raphanus sativus var.
longipinnatus L.
H. Bailey (Daikon Group) | Japan | Improved
cultivar |
| JP27237 | NAGAGATA HARUWAKA | Raphanus sativus var.
longipinnatus L.
H. Bailey (Daikon Group) | Japan | Traditional
cultivar |
| JP73395 | RAVE BLANCHE | Raphanus sativus var.
longipinnatus L.
H. Bailey (Daikon Group) | MAURITIUS | Traditional
cultivar |
| JP73397 | SAKSA | Raphanus sativus var.
longipinnatus L.
H. Bailey (Daikon Group) | Unknown | Unknown |
| JP76693 | OZENA DAIKON | Raphanus sativus var.
longipinnatus L.
H. Bailey (Daikon Group) | Japan | Traditional
cultivar |
| JP76696 | KARAMI DAIKON | Raphanus sativus var.
longipinnatus L.
H. Bailey (Daikon Group) | Japan | Traditional
cultivar |
| RA128 | White Icicle | Raphanus sativus convar. sativus
(Small radish group) | USA | Improved
cultivar |
| RA133 | Triumph | Raphanus sativus convar. sativus
(Small radish group) | Switzerland | Improved
cultivar |
| RA134 | Koral | Raphanus sativus convar. sativus
(Small radish group) | Czechoslovakia | Improved
cultivar |
| RA148 | Unknown | Raphanus sativus convar. sativus
(Radish group) | Afghanistan | Traditional
cultivar |
| RA157 | Giant Japanese
Radish | Raphanus sativus convar. sativus
(Radish group) | Portugal | Improved
cultivar |
| RA165 | Sofijskaja
Prevazhodna | Raphanus sativus convar. sativus
(Small radish group) | Bulgaria | Improved
cultivar |
| RA166 | Unknown | Raphanus sativus convar. sativus
(Small radish group) | Armenia | Traditional
cultivar |
| RA199 | Husveti Rozsa | Raphanus sativus convar. sativus
(Radish group) | Hungary | Improved
cultivar |
| RA213 | Unknown | Raphanus sativus convar. sativus
(Radish group) | Libya | Traditional
cultivar |
| RA229 | Baeg Am Mu | Raphanus sativus convar. sativus
(Radish group) | North Korea | Improved
cultivar |
| RA280 | Unknown | Raphanus sativus convar. sativus
(Radish group) | Kazakhstan | Traditional
cultivar |
| RA298 | chjatag bajcaa
(mong.) | Raphanus sativus convar. sativus
(Radish group) | Mongolia | Traditional
cultivar |
| RA299 | Unknown | Raphanus sativus convar. sativus
(Small radish group) | Ukraine | Traditional
cultivar |
| RA353 | Fijil Aswad | Raphanus sativus convar. sativus
(Radish group) | Iraq | Traditional
cultivar |
| RA365 | Unknown | Raphanus sativus convar. sativus
(Radish group) | Romania | Traditional
cultivar |
| RA41 | Unknown | Raphanus sativus convar. sativus
(Radish group) | Greece | Traditional
cultivar |
| RA420 | Unknown | Raphanus sativus L. subp.
Raphanistrum (Moretti Ex Dc.) | Belgium | Wild |
| RA482 | Certina | Raphanus sativus convar. sativus
(Small radish group) | Germany | Improved
cultivar |
| RA502 | Janosnapi | Raphanus sativus convar. sativus
(Small radish group) | Hungary | Improved
cultivar |
| RA522 | Saya Daikon | Raphanus sativus convar. caudatus
(L.f.) Pistrick | India | Improved
cultivar |
| RA751 | Unknown | Raphanus sativus convar. sativus
(Small radish group) | Azerbaijan | Traditional
cultivar |
| RA801 | unknown | Raphanus raphanistrum L. subsp.
raphanistrum x R. raphanistrum
L. ssp. landra (Moretti ex DC.)
Bonnier et Layens s.l. | Greece | Wild |
| RA829 | Unknown | Raphanus sp. | North Korea | Unknown |
| RA832 | Unknown | Raphanus sativus convar. sativus
(Small radish group) | Spain | Unknown |
시료의 추출
82 계통의 종자는 직파 후 하우스에서 재배하였고, 약 8주 후에 계통 별로 3 개체를 선발하여 생 무를 채취한 후 총 유리당 함량을 분석하였다. 생 무는 동결건조 후 분쇄하였고, 샘플 2 g을 15 ㎖ 튜브에 넣은 후 5% trichloroacetic acid (TCA) 10 ㎖을 첨가하고 1분간 혼합한 후 4℃에서 1시간동안 반응시켰다. 반응액은 10000g, 4℃에서 15분간 원심 분리하여 회수한 상층액을 여과한 후 유리당 함량 분석에 사용하였다.
유리당 함량 분석
총 유리당 함량을 측정하기 위하여 CarboPac™(PAI (4 * 250 ㎜), P/N 35391, 미국) 칼럼을 장착한 고속 액체 크로마토그래피 시스템을 사용하여 분석을 실시하였다. 이동상은 150 mM NaOH (solvent A)와 150 mM NaOH, 600 mM sodium acetate (solvent B)가 혼합된 두 종류의 용매를 유속 1 ㎖·min-1로 사용하였다. 표준물질은 포도당(glucose), 과당(fructose), 자당(sucrose), 그리고 엿당(maltose)을 증류수와 혼합하여 사용하였다. 시료 내의 총 유리당은 표준 용액과 시간을 비교하여 확인한 후 표준검량곡선을 작성하여 유리당 함량을 산출하였다.
결과 및 고찰
무 유전자원의 외형적 특성
한국, 일본, 그리고 독일의 유전자원센터에서 보유중인 82 계통의 무 유전자원을 하우스에서 재배하여 외형적 형질의 특성을 관찰하였다. 개화시기를 포함한 생리학적 특성과 잎, 뿌리, 꽃의 크기, 형태 및 색깔 등의 형태학적 특성에서 계통에 따른 차이가 관찰되었다. 꽃은 흰색(IT110840), 연보라색(JP26959), 자주색(IT119238), 그리고 분홍색(RA213)의 네 가지로 구분할 수 있었고, 뿌리의 경우 흰색(JP27211)과 자주색(IT262007)의 두 가지로 크게 구분할 수 있었다(Fig. 1). 식물에서의 꽃 색은 다양한 수분 매개자를 유도하는 작용을 하기 때문에 육종적인 측면에서 종자의 생산성을 좌우하는 중요한 형질이다(Irwin and Strauss, 2005). 또한 요리 재료로 사용되는 뿌리 조직의 다양한 형태 및 색깔은 소비자 성향에 따른 외형적 품질지표로 중요할 뿐만 아니라 식물 색소 형성에 관여하는 것으로 알려진 안토시아닌과 같은 식물의 이차대사산물의 함량과도 매우 밀접한 관련이 있다(Giusti et al., 1998; Kim et al., 2017). 본 연구에서 수집한 82 계통의 무 유전자원은 꽃과 뿌리 조직의 색소 형질 외에도 개화 시기, 조직 경도 및 잎의 형태 등에서 다양한 변이가 관찰되었다. 현재, 82 계통이상의 무 유전자원을 대상으로 광범위한 형질 조사가 진행 중이며, 이러한 결과를 토대로 우수 계통을 선발, 육성한다면, 고품질의 무 품종 개발에 기여할 수 있을 것이다.

Fig. 1.
Color variation of flowers and roots in 82 accessions of radish.
총 유리당 함량
식물의 중요한 탄소 공급원인 유리당의 총 함량을 분석하기 위해서 무 유전자원 82 계통의 뿌리 조직을 채취한 후 고속 액체 크로마토그래피를 이용하여 분석을 실시하였다(Table 2). 포도당은 평균 14.07±5.48 ㎎/g의 함량을 보였고, IT216713이 3.26±0.32 ㎎/g의 가장 낮은 함량을 보인 반면에, RA280의 경우는 IT 216713 보다 약 10배 높은 30.39±1.63 ㎎/g의 높은 포도당 함량을 보였다. 평균 7.97±4.08 ㎎/g의 함유량을 가진 과당은 최소 0.91±0.29 ㎎/g에서 최대 18.43±2.01 ㎎/g까지 다양한 분포를 보였다. 반면에 자당과 엿당의 경우, 각각 평균 2.974±4.261 ㎎/g과 0.515±1.210 ㎎/g의 대체적으로 낮은 함유량을 보였고, 자당이 분석되지 않을 정도로 낮은 함량을 가지는 21 계통이 존재하였고, 절반 이상의 계통인 48계통에서는 엿당이 분석되지 않았다. 특히 자당이 분석되지 않은 21 계통들은 RA298을 제외한 20 계통 모두에서 엿당 또한 분석되지 않았다. 총 유리당 함량의 경우 RA502에서 46.68 ㎎/g으로 가장 높은 함량을 보였고, RA134 계통은 5.64±2.97 ㎎/g으로 가장 낮은 유리당을 함유하고 있었다. 또한 총 유리당 함량에 대한 각 유리당 함량의 비율을 분석한 결과, 포도당은 56.92%로 가장 높은 비율을 차지하였고, 엿당의 경우 2.25%로 가장 낮은 함유율을 나타내었다(Fig. 2, total). 본 실험의 결과, 모든 유전자원에서 자당과 엿당의 함유량은 포도당과 과당에 비해 매우 낮은 수준으로 유지되고 있었으며, 이와 동일한 연구 결과는 선행된 연구에서도 보고되었다(Hara et al., 2003; 2011).
Table 2. Sugars content (mg/g) in 82 accessions of R. sativus
| Accession | Glucose | Fructose | Sucrose | Maltose | Total |
| CR1787 | 17.18±1.79z | 12.82±1.76z | 1.65±0.33z | 0.62±0.06z | 32.27±3.38z |
| CR2970 | 21.19±1.06 | 2.66±0.24 | 3.63±1.03 | 0.00±0.00 | 27.47±0.97 |
| CR3472 | 12.27±1.12 | 9.77±1.04 | 1.45±0.35 | 0.00±0.00 | 23.50±1.92 |
| CR499 | 10.65±1.16 | 7.42±0.82 | 2.19±0.22 | 0.00±0.00 | 20.26±1.77 |
| CR504 | 18.10±3.30 | 5.52±0.35 | 5.75±1.44 | 0.00±0.00 | 29.37±4.85 |
| IT 102547 | 15.19±1.45 | 4.87±1.09 | 3.01±0.67 | 0.00±0.00 | 23.07±2.57 |
| IT110840 | 14.92±1.13 | 12.58±1.06 | 0.00±0.00 | 0.00±0.00 | 27.50±1.29 |
| IT 110843 | 18.41±1.09 | 12.09±0.32 | 5.76±0.54 | 2.09±0.61 | 38.34±2.10 |
| IT 113530 | 18.14±3.99 | 11.15±2.86 | 0.91±0.29 | 0.00±0.00 | 30.20±7.10 |
| IT 119228 | 20.90±1.28 | 16.49±1.18 | 4.05±0.56 | 0.21±0.02 | 41.65±2.94 |
| IT 119235 | 16.93±1.78 | 8.95±1.20 | 1.49±0.30 | 0.00±0.00 | 27.36±3.20 |
| IT 119238 | 20.00±2.35 | 18.43±2.01 | 2.39±0.08 | 0.24±0.03 | 41.06±4.45 |
| IT 119252 | 15.82±2.96 | 0.91±0.29 | 0.00±0.00 | 0.00±0.00 | 16.73±3.25 |
| IT 119279 | 21.79±4.44 | 3.22±1.03 | 3.45±0.94 | 0.16±0.06 | 28.61±6.17 |
| IT 119281 | 13.90±0.20 | 10.39±0.78 | 0.00±0.00 | 0.00±0.00 | 24.29±0.61 |
| IT 119282 | 14.70±3.23 | 5.72±0.98 | 0.00±0.00 | 0.00±0.00 | 20.42±3.81 |
| IT 119287 | 22.34±0.29 | 17.21±0.49 | 3.37±0.14 | 0.43±0.02 | 43.34±0.74 |
| IT 119294 | 11.42±1.40 | 6.78±0.72 | 0.00±0.00 | 0.00±0.00 | 18.19±2.11 |
| IT 119299 | 19.49±0.87 | 9.78±0.74 | 5.48±0.24 | 0.00±0.00 | 34.74±1.34 |
| IT 119304 | 22.37±0.89 | 3.58±0.46 | 0.52±0.46 | 0.00±0.00 | 26.47±0.10 |
| IT 119318 | 11.11±2.44 | 9.70±1.98 | 1.45±0.78 | 0.00±0.00 | 22.27±5.19 |
| IT 119319 | 11.74±0.72 | 9.67±0.46 | 1.58±0.14 | 0.49±0.05 | 23.48±1.30 |
| IT 136490 | 12.68±1.41 | 11.09±1.54 | 2.62±2.80 | 0.00±0.00 | 26.39±0.13 |
| IT 138209 | 4.71±0.44 | 2.70±0.33 | 6.80±1.11 | 0.00±0.00 | 14.21±1.66 |
| IT 180726 | 9.44±0.22 | 6.59±0.15 | 0.00±0.00 | 0.00±0.00 | 16.02±0.35 |
| IT 186722 | 11.49±0.71 | 5.33±0.71 | 0.00±0.00 | 0.00±0.00 | 16.82±0.37 |
| IT 188101 | 16.74±1.10 | 10.40±1.33 | 5.81±1.07 | 1.06±0.07 | 34.02±2.81 |
| IT 203302 | 9.70±2.74 | 5.95±1.34 | 0.43±0.13 | 0.00±0.00 | 16.07±4.14 |
| IT 209972 | 20.87±5.85 | 14.17±4.00 | 1.38±0.23 | 0.00±0.00 | 36.42±10.05 |
| IT 215053 | 22.76±0.45 | 5.06±0.90 | 0.29±0.05 | 0.58±0.03 | 28.68±1.28 |
| IT 216713 | 3.26±0.32 | 1.32±0.16 | 10.81±0.96 | 1.04±0.18 | 16.42±1.61 |
| IT 250744 | 14.82±1.46 | 7.05±1.33 | 2.37±0.41 | 0.00±0.00 | 24.24±2.80 |
| IT 250748 | 17.35±4.05 | 10.94±2.65 | 1.49±0.64 | 0.34±0.18 | 30.12±7.50 |
| IT 250766 | 8.29±0.29 | 4.95±1.04 | 19.41±0.96 | 0.00±0.00 | 32.65±1.06 |
| IT 250769 | 12.64±0.44 | 3.19±0.58 | 0.44±0.05 | 0.00±0.00 | 16.27±1.06 |
| IT 250770 | 15.24±1.17 | 12.28±0.97 | 1.61±0.97 | 0.99±0.93 | 30.13±3.07 |
| IT 250780 | 5.09±0.24 | 3.65±0.37 | 0.81±0.10 | 0.00±0.00 | 9.55±0.61 |
| IT 250793 | 23.41±0.85 | 8.44±0.41 | 6.65±1.58 | 0.92±0.08 | 39.42±2.27 |
| IT 261002 | 5.13±0.63 | 2.22±0.35 | 14.54±0.87 | 2.91±2.11 | 21.89±1.80 |
| IT 261947 | 16.12±0.38 | 13.03±0.66 | 0.00±0.00 | 0.00±0.00 | 29.15±0.92 |
| IT 261997 | 15.37±1.16 | 10.37±1.03 | 1.22±1.02 | 0.41±0.05 | 27.38±1.29 |
| IT 261999 | 6.45±1.81 | 2.70±0.53 | 0.00±0.00 | 0.00±0.00 | 9.15±2.29 |
| IT 262007 | 13.24±1.21 | 11.70±0.92 | 0.46±0.04 | 0.00±0.00 | 25.40±2.12 |
| IT 262017 | 13.16±1.00 | 9.58±0.69 | 0.00±0.00 | 0.00±0.00 | 22.74±1.68 |
| IT 263949 | 17.85±1.60 | 4.55±2.01 | 1.98±0.49 | 0.22±0.05 | 24.59±1.02 |
| IT 276363 | 10.04±1.61 | 6.72±0.76 | 21.35±1.82 | 0.73±0.22 | 38.83±4.31 |
| IT 276488 | 15.47±1.40 | 11.44±2.92 | 0.23±0.01 | 0.41±0.09 | 27.55±3.86 |
| IT 277626 | 8.64±0.48 | 8.08±0.45 | 1.67±0.60 | 0.19±0.10 | 18.57±1.31 |
| IT 278270 | 4.82±2.12 | 2.00±0.98 | 0.00±0.00 | 0.00±0.00 | 6.82±3.10 |
| IT 291148 | 9.77±1.21 | 7.71±0.70 | 1.50±0.62 | 0.00±0.00 | 18.97±2.22 |
| JP26959 | 16.20±0.51 | 12.82±0.33 | 0.62±0.43 | 0.28±0.05 | 29.91±1.15 |
| JP27208 | 11.18±1.41 | 8.38±0.82 | 0.00±0.00 | 0.00±0.00 | 19.56±1.93 |
| JP27211 | 18.20±0.68 | 13.65±0.43 | 0.85±0.26 | 0.00±0.00 | 32.70±1.07 |
| JP27237 | 10.34±0.24 | 8.16±0.14 | 7.08±1.16 | 0.51±0.08 | 26.09±1.09 |
| JP73395 | 10.08±1.42 | 6.54±1.11 | 0.00±0.00 | 0.00±0.00 | 16.62±2.51 |
| JP73397 | 14.89±0.99 | 6.18±0.18 | 0.00±0.00 | 0.00±0.00 | 21.07±1.16 |
| JP76693 | 4.22±1.03 | 2.82±0.67 | 7.35±2.11 | 6.61±3.91 | 14.39±3.80 |
| JP76696 | 10.93±0.63 | 7.21±0.22 | 5.73±2.09 | 0.96±0.74 | 24.84±3.63 |
| RA128 | 11.07±0.33 | 2.21±0.19 | 0.00±0.00 | 0.00±0.00 | 13.28±0.25 |
| RA133 | 6.64±0.57 | 4.41±0.66 | 0.91±0.22 | 2.01±1.46 | 11.97±0.96 |
| RA134 | 3.28±1.72 | 2.36±1.25 | 0.00±0.00 | 0.00±0.00 | 5.64±2.97 |
| RA148 | 6.59±1.29 | 5.13±0.88 | 0.00±0.00 | 0.00±0.00 | 11.72±2.17 |
| RA157 | 11.16±0.69 | 5.16±0.55 | 15.72±0.32 | 0.67±0.07 | 32.70±0.88 |
| RA165 | 20.05±5.05 | 3.86±0.60 | 0.46±0.27 | 0.33±0.20 | 24.71±5.61 |
| RA166 | 16.81±1.11 | 14.32±0.41 | 4.42±1.45 | 0.27±0.11 | 35.82±2.36 |
| RA199 | 10.19±0.51 | 6.72±0.37 | 0.21±0.06 | 0.00±0.00 | 17.12±0.93 |
| RA213 | 10.31±2.08 | 7.46±1.76 | 0.00±0.00 | 0.00±0.00 | 17.77±3.82 |
| RA229 | 14.02±1.06 | 3.07±0.24 | 3.80±1.83 | 0.00±0.00 | 20.88±1.70 |
| RA280 | 30.39±1.63 | 6.55±0.37 | 0.00±0.00 | 0.00±0.00 | 36.94±1.53 |
| RA298 | 6.44±1.72 | 5.60±1.24 | 0.00±0.00 | 5.67±7.82 | 17.71±10.63 |
| RA299 | 13.02±0.88 | 3.76±0.53 | 4.45±2.40 | 0.00±0.00 | 21.24±2.82 |
| RA353 | 13.34±1.04 | 8.48±0.55 | 1.57±0.54 | 0.00±0.00 | 23.39±1.51 |
| RA365 | 23.09±3.25 | 6.52±0.49 | 9.31±0.88 | 0.54±0.07 | 39.46±4.19 |
| RA41 | 12.60±0.62 | 1.56±0.13 | 3.63±0.37 | 1.46±0.09 | 19.26±1.11 |
| RA420 | 13.83±0.21 | 10.53±0.45 | 1.11±0.50 | 0.00±0.00 | 25.47±0.56 |
| RA482 | 13.73±0.75 | 10.92±0.66 | 0.37±0.11 | 0.00±0.00 | 25.01±1.45 |
| RA502 | 26.62±1.10 | 7.57±0.44 | 10.07±0.23 | 2.42±0.12 | 46.68±1.71 |
| RA522 | 16.05±1.76 | 14.54±1.87 | 7.47±0.73 | 0.26±0.03 | 38.32±4.17 |
| RA751 | 16.91±1.02 | 11.46±0.65 | 1.83±0.38 | 5.68±1.96 | 35.88±3.86 |
| RA801 | 14.72±1.28 | 12.40±1.88 | 0.00±0.00 | 0.00±0.00 | 27.12±3.12 |
| RA829 | 18.43±0.63 | 13.61±1.82 | 2.77±1.21 | 0.49±0.05 | 35.29±1.35 |
| RA832 | 15.41±0.55 | 11.59±0.12 | 2.10±0.27 | 0.00±0.00 | 29.11±0.41 |
| 14.07±5.48 | 7.91±4.08 | 2.97±4.26 | 0.52±1.21 | 25.33±8.86 |
zThe means indicate the average of triplicate measurements in three independent biological replicates.
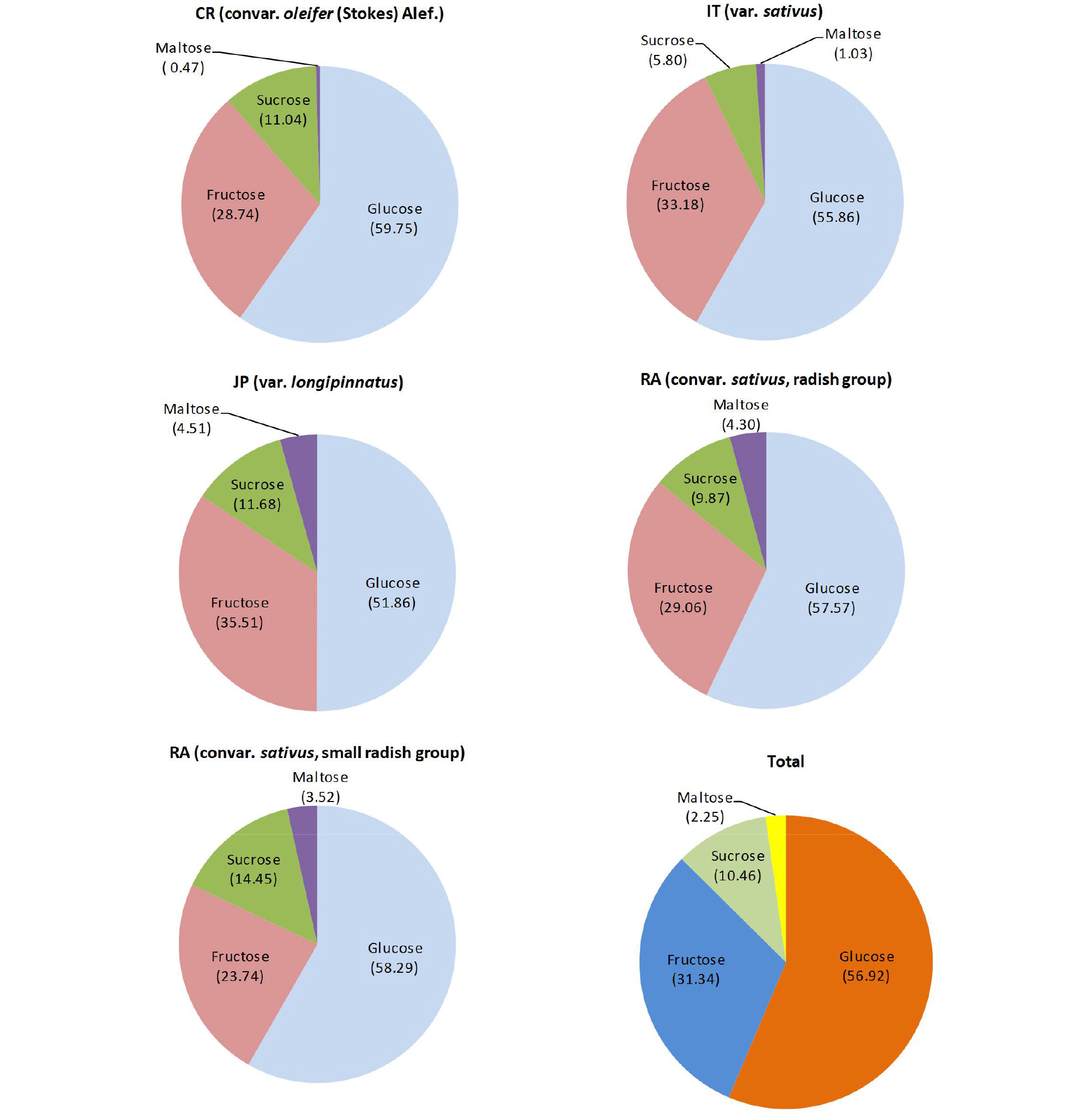
Fig. 2.
Ratio of the four sugars in total sugars content by subspecies in 82 accessions of radish.
유전형에 따른 유리당 함량 분석
일본, 독일, 그리고 한국에서 수집한 무 유전자원을 아종(subspecies)에 따라 분리하여 총 유리당 함량을 분석하였다. 일부 아종을 제외한 총 5개 아종에 속하는 75 계통들에 관한 총 유리당 함량을 분석한 결과는 Table 3과 같다. 농촌진흥청, 유전자원센터에서 보유하고 있는 IT 계통(var. sativus)은 우리나라를 포함한 다양한 나라를 기원으로 하는 무의 유전자원이며, 일본에서 보유중인 JP 계통(var. longipinnatus)은 영어로 daikon, 우리말로는 왜무로 불리는 희고 긴 무로 주로 단무지의 재료로 사용된다. 독일에서 수집한 CR 계통(convar. oleifer (Stokes) Alef.)은 oilseed radish로 불리며 주로 가축의 사료로 사용되며, RA계통(convar. sativus)은 서양 붉은 순무로 불리며 주로 유럽에서 재배되며 크기가 작고 생육기간이 짧으며 붉고 둥근 모양을 띄는 small radish group과 일반적인 흰 무의 크기를 가진 radish group으로 분류할 수 있었다. 총 유리당 함량의 평균은 최소 23.148 ㎎/g에서 최대 26.574 ㎎/g으로 아종에 따른 총 유리당 함량은 큰 차이를 보이지 않았다. CR 계통들은 평균 15.878 ㎎/g의 가장 높은 포도당 함량을 보였고, 과당은 IT 계통, 자당은 RA의 small radish group, 그리고 엿당은 RA의 radish group에서 가장 높은 평균 유리당 함량을 보였다. 또한, 총 유리당 함량에 대한 각 유리당 함량의 비율을 분석한 결과, 포도당, 과당, 자당, 엿당 순으로 높은 함유율을 보였고, 포도당과 자당의 경우 평균 함량이 높은 CR과 RA의 small radish group 계통들에서 59.75%와 14.45%의 가장 높은 함유율이 관찰되었다. 반면에, 과당과 엿당의 경우에서는 JP 계통에서 가장 높은 함유율을 보였다(Fig. 2). 십자화과 식물에서 영양성분의 함량은 식물의 유전적 요인에 영향을 받는 것으로 알려져 있다(Murtaza et al., 2005). 최근에는 십자화과 식물인 배추에서 글루코시놀레이트 함량을 아종별로 분류하여 분석한 결과, 아종에 따른 글루코시놀레이트 함량의 차이를 확인할 수 있었다(Seo et al., 2017). 본 실험에서는 무의 아종에 따른 총 유리당 함량에는 큰 차이를 보이지 않았으나 유리당의 종류에 따른 함유 율에는 차이가 관찰되었다. 무의 유리당 함량에 영향을 미치는 유전적 요인을 증명하기 위해서는 광범위한 형질 조사와 함께 수집된 유전자원의 유전적 분석을 통한 계통 분류를 실시하여 유리당 함량과의 상관관계를 명확하게 분석할 필요가 있을 것이다.
Table 3. Total and individual sugars level (mg/g) in roots of 75 accessions belonging to five genotypes of R. sativus
| Genotype | Accession
number | Glucose | Fructose | Sucrose | Maltose | Total |
CR (convar. oleifer (Stokes)
Alef.) | | | | | | |
| Meanz | 5 | 15.878 | 7.638 | 2.934 | 0.124 | 26.574 |
| Range | | 10.65-21.19 | 2.66-12.82 | 1.45-3.63 | 0-0.62 | 20.26-32.27 |
| IT (var. sativus) | 41 | | | | | |
| Meanz | | 14.446 | 8.58 | 1.5 | 0.266 | 25.859 |
| Range | | 4.71-22.76 | 0.91-17.21 | 0-21.35 | 0-2.09 | 6.82-43.34 |
| JP (var. longipinnatus) | 8 | | | | | |
| Meanz | | 12.005 | 8.22 | 2.704 | 1.045 | 23.148 |
| Range | | 4.22-18.2 | 6.18-13.65 | 0-7.35 | 0-6.61 | 14.39-32.7 |
RA (convar. sativus, radish
group) | 10 | | | | | |
| Meanz | | 14.354 | 7.246 | 2.461 | 1.071 | 24.934 |
| Range | | 3.28-26.62 | 2.21-14.32 | 0-10.07 | 0-5.68 | 5.64-46.68 |
RA (convar. sativus, small
radish group) | 10 | | | | | |
| Meanz | | 13.813 | 5.625 | 3.424 | 0.834 | 23.695 |
| Range | | 6.44-30.39 | 3.07-8.48 | 0-15.72 | 0-5.67 | 11.72-39.46 |
zThe means indicate the average of subspecies with the average of triplicate measurements in three independent biological replicates.
자원의 구분에 따른 유리당 함량 분석
82 계통의 무 유전자원에서 정확한 자원의 구분이 어려운 8 계통을 제외한 74 계통은 야생종, 야생 근연종, 재래종, 그리고 육성품종으로 구분할 수 있었다. 총 유리당 함량의 평균은 최소 24.64 ㎎/g에서 최대 25.63 ㎎/g으로 큰 차이를 보이지 않았다(Table 4). 포도당과 과당은 야생종에서 각각 14.58 ㎎/g과 9.27 ㎎/g으로 가장 높은 함유량을 보인 반면, 자당과 엿당은 재래종에서 4.11 ㎎/g과 0.93 ㎎/g으로 가장 높은 함유량이 관찰되었다. 또한 각 유리당의 함유 비율을 자원에 따라 분석한 결과, 함유량과 마찬가지로 포도당과 과당은 57.81%와 36.74%로 야생종에서 가장 높은 함유 율을 나타내었고, 자당과 엿당은 재래종에서 각각 16.62%와 3.78%로 가장 높은 함유 율을 보였다(Fig. 3). 일반적으로, 과당은 포도당과 자당에 비해 단맛에 영향을 주는 것으로 알려져 있다. 같은 농도의 포도당, 과당, 자당에 대하여 단맛을 측정한 결과, 과당, 자당, 포도당 순으로 단맛에 영향을 미치는 것을 확인할 수 있었다(Mahawanich and Schmidt, 2004; Zamora et al., 1998). 본 실험의 결과, 야생 근연종인 IT119238은 가장 높은 과당 함량을 보였고, 야생종은 야생 근연종, 재래종, 그리고 육성품종과 비교하여 평균 과당 함량이 가장 높게 관찰되었다. 재래종과 육성품종을 비교하였을 때, 총 유리당 함량은 재래종 24.72 ㎎/g, 육성종 24.64 ㎎/g으로 큰 차이를 보이지 않았으나, 과당의 비율은 재래종 27.84%에서 육성종 33.6%로 증가하였고, 자당의 비율은 재래종 16.62%에서 육성종 9.09%로 오히려 감소하였다. 이러한 결과는, 선발 육종과정에서 품질지표인 단맛이 높은 계통을 반복적으로 선별하여, 자당의 비율은 감소하고, 단맛에 크게 영향을 미치는 과당의 비율이 증가한 육성 품종이 선발된 것으로 추정된다. 또한 당의 종류에 따른 동물의 비만도를 측정한 결과, 자당은 과당에 비하여 쥐의 비만을 초래한다는 연구 결과가 보고되었다(Glendinning et al., 2010). 따라서 샐러드 및 김치를 포함한 다양한 요리 재료로 활용하기 위한 무의 선발 육종에서는 총 유리당 함량에서 차지하는 과당의 높은 함유 율이 매우 중요한 선발 기준이 될 수 있을 것이다.
Table 4. Total and individual sugars level (mg/g) in roots of 74 accessions belonging to various biological status of R. sativus
| Cultivar types | Accession
number | Glucose | Fructose | Sucrose | Maltose | Total |
| Wild |
| Meanz | 3 | 14.58 | 9.27 | 1.37 | 0.00 | 25.22 |
| Range | | 13.83-15.19 | 4.87-12.4 | 0.00-3.01 | 0.00 | 23.07-27.12 |
| Wild relatives |
| Meanz | 20 | 14.56 | 7.70 | 3.19 | 0.34 | 25.63 |
| Range | | 3.26-22.37 | 0.91-18.43 | 0.00-14.54 | 0.00-2.91 | 14.21-43.34 |
| Traditional cultivar |
| Meanz | 25 | 13.06 | 6.88 | 4.11 | 0.93 | 24.72 |
| Range | | 4.22-30.39 | 2.00-14.32 | 0.00-21.35 | 0.00-6.61 | 6.82-39.46 |
| Improved cultivar |
| Meanz | 26 | 13.93 | 8.15 | 2.24 | 0.40 | 24.64 |
| Range | | 3.28-26.62 | 2.26-14.54 | 0.00-15.72 | 0.00-2.42 | 5.64-46.68 |
zThe means indicate the average of triplicate measurements in three independent biological replicates.
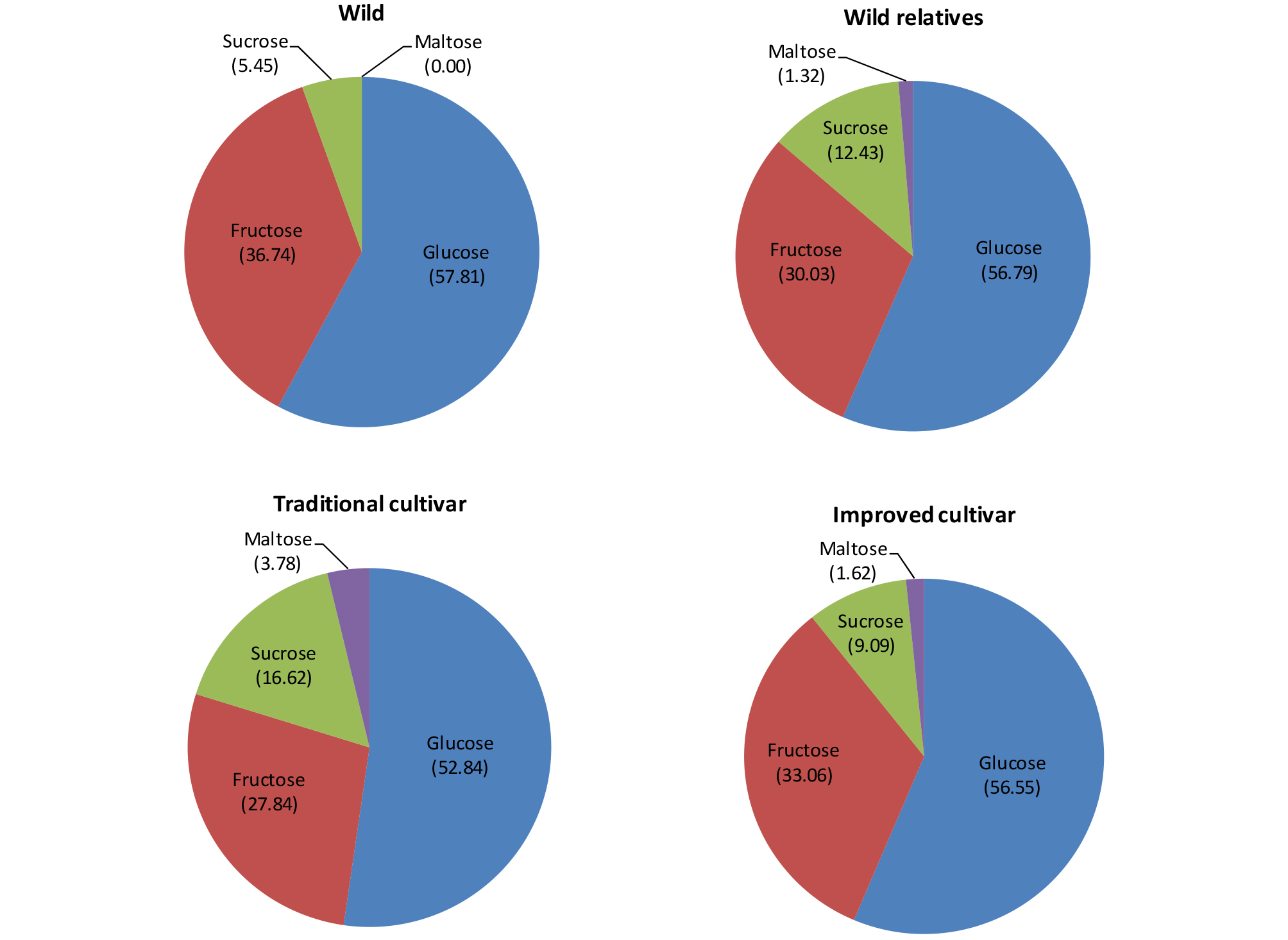
Fig. 3.
Ratio of the four sugars in total sugars content by biological status in the 74 accessions of radish.
식물 자원에서 야생종, 야생 근연종 및 재래종 자원은 유전적 다양성과 함께 농업적으로 유용한 특성을 보유하고 있는 것으로 알려져 있다(Ellstrand and Marshall, 1985). 무의 유전자원에서 유리당 함량을 분석한 결과, 높은 유리당 함량을 보유한 야생 근연종과 재래종 계통들이 관찰되었다. 또한 야생종과 재래종은 유리당 종류에 따라 육성 품종과 구분되는 함유 율을 보였다. 따라서 82 계통의 유전자원을 대상으로 광범위한 형질조사를 실시한다면, 품질 지표로 사용되는 단맛을 포함하여 농업적으로 우수한 형질을 보유한 품종의 개발이 가능할 것으로 기대된다.
식물에서 유리당의 함량은 단맛에 영향을 미치는 요인으로 작용할 뿐만 아니라 환경적 변화에 대응하는 조절 메커니즘에도 관여하는 것으로 알려져 있다(Dobrenel et al., 2013). 본 연구결과는 무의 다양한 형질특성을 가진 유전자원 82 계통에 관한 총 유리당 함유량을 분석함으로써 고당도 무 품종 및 환경 변화에 내성을 가진 우수 계통의 선발뿐만 아니라 무의 유리당 합성에 관여하는 메커니즘을 규명하기 위한 기초 정보를 제공할 수 있을 것으로 사료된다.
적요
십자화과 식물에 속하는 무는 전세계적으로 재배되는 중요한 뿌리 채소작물이다. 본 연구에서는 다양한 형질을 가진 무 유전자원 82 계통을 대상으로 총 유리당 함량을 분석하였다. 일본, 독일, 그리고 한국에서 보유중인 82 계통의 유전자원은 크게 5 아종을 포함하고 있으며, 야생형, 야생 근연종, 재래종 그리고 육성품종으로 구분된다. 포도당, 과당, 자당 그리고 엿당을 분석하여 총 유리당 함량을 측정한 결과, 평균 25.33 ㎎/g, 5.64-46.68 ㎎/g의 분포도를 나타내어 계통에 따라 총 유리당 함량에 차이를 확인할 수 있었다. 또한 총 유리당 함량에 대하여 포도당이 56.92%로 가장 높은 유리당 함유율을 보였고, 과당(31.34%), 자당(10.46%), 그리고 엿당(2.25%)의 순으로 높은 함유 율을 보였다. 5 아종에서 유전형에 따른 총 유리당 함량의 차이는 관찰되지 않았으나, 아종에 따른 유리당 함유 율의 차이는 확인할 수 있었다. 또한 야생종과 야생 근연종 그리고 재래종 무는 육성 품종과 비교하여 평균 총 유리당 함량에서는 큰 차이를 보이지 않았으나, 당의 종류별 함유 율을 비교한 결과 야생종과 재래종에서 높은 함유 율을 확인할 수 있었다. 본 실험의 결과는 후속 연구를 통한 고품질 무의 개발 및 무의 유리당 생합성 관련 연구를 위한 기초정보를 제공할 수 있을 것이다.







