서 언
콩은 단백질(35~40%)과 지방(15~20%)의 주요 공급원으로서 한국뿐 아니라 전 세계적으로 재배되는 주요 작물 중에 하나이다. 한국에서는 오래 전부터 콩의 용도에 따라 장류 및 두부, 나물, 밥밑콩, 풋콩 및 단기성 콩 등 다양한 형태로 이용하여 왔으며 최근에는 건강식품 및 화장품 원료 등 산업화 소재로도 이용하고 있다. 현재까지 국내에는 180여 품종이 국립종자원에 품종보호 등록이 되어 있으며 이 외에도 10여 품종이 품종등록을 위해 재배심사 중에 있다(http://www.seed.go.kr/, 2019년 8월 기준).
가공용 등 콩의 국내 연간 수요량은 2016년 기준으로 약 36만 톤 정도인데 반해 국산 자급율은 약 20% (7.5만 톤)에 불과하여 대부분 미국, 브라질, 중국, 아르헨티나 등에서 수입되고 있다. 가격은 국내산이 수입산에 비해 4배 비싸지만 소비자의 기호는 국내산에 대한 신뢰가 높아 일부 수입콩은 국산으로 둔갑되어 판매되기도 한다. 이로 인해 소비자들의 피해가 커지고 국산콩에 대한 신뢰 감소, 생산자와 육성가의 권리 침해 등을 야기하여 국산 품종보호를 위한 품종판별 기술 개발이 요구되고 있다. 우리나라는 1995년 종자산업법 제정과 함께 품종보호제도를 도입하여 신품종 육성가의 권리를 법적으로 보호하고 있는데 이를 위해 품종등록 단계에 신품종의 신규성, 구별성, 균일성, 안전성 등의 요건을 갖추어야 한다.
신품종의 구별성은 주로 심사단계에서 대조품종에 대한 잎모양, 종실 크기, 종피색, 배축색 등 형태적인 차이에 근거를 두고 있으나 농업형질은 외부 환경변화에 영향을 받을 수 있다. 최근에는 국제신품종보호동맹(International Union for the Protection of New Varieties of Plants, UPOV)에서 신품종 구별성을 검증하기 위해 DNA 마커의 활용을 검토하고 있다(Kim et al., 2011). DNA 분자마커를 이용한 품종판별은 작목별로 활발하게 개발되고 있으며 콩에서도 SSR (simple sequence repeat), STS (sequence tagged sites), CAPS (cleaved amplification polymorphic sequence), SNP (single nucleotide polymorphism), InDel (insertion/deletion) 등의 마커가 품종판별과 유전 육종연구를 위해서 보고되고 있다(Hwang et al., 2009; Lee et al., 2017; Liu et al., 2012; Moghaddam et al., 2014; Mwangi et al., 2019; Song et al., 2010). SSR 마커의 경우 다형성이 높기 때문에 품종판별 마커로 주로 이용되고 있으나 분석방법이 까다롭고 시간이 많이 걸리는 단점이 있으며, STS 마커의 경우 분석이 간단하나 유전변이가 높지 않으며, SNP 변이는 유전체에 매우 고르게 다량 분포하고 있으나 분석에 필요한 특정 장비를 요구하는 경우가 많다.
최근 유전체 분석 기술이 발달하여 유전정보를 확인하는 과정이 용이하게 되어(Nguyen et al., 2019), 연구집단 유전체정보(resequencing, RNA sequencing, GBS 등)로부터 다양성을 분석하고 DNA 마커를 개발하는 효율성이 높아졌다(Agarwal et al., 2008; Hyten et al., 2010; Kim et al., 2010; Kim et al., 2019; Korir et al., 2013; Lee et al., 2018; Yaqi, 2018). 유전체 기반 마커 탐색 방법은 SSR (Simple Sequence Repeat) 마커를 이용하여 연구집단의 다양성을 추정하던 방법보다 정확하고 많은 수의 마커를 제작할 수 있다. 유전체 기반 마커 개발은 품종판별(Heo et al., 2017), 벼멸구저항성 유전자 탐색(Ji et al., 2018) 등 분자육종 연구, 육종소재 및 산업화 소재로서 자원을 개발하기 위한 유전자원 다양성분석 등으로 최근 널리 활용되고 있다.
기존 마커의 장단점을 보완하여 Sohn et al. (2017a)은 콩 전장유전체재분석(resequencing) 정보로부터 실험실에서 간단하게 분석할 수 있는 공우성(co-dominant)의 InDel 마커를 개발하였다. 6개 콩 품종의 유전체를 비교하여 한국 콩 품종의 변이밀집영역(dense Variation Block, dVB)을 분석하고 이 영역에서 InDel 마커 202종을 개발하여 국내 등록품종 등 149품종에 대한 유전형을 분석하였다. dVB에 기반하여 개발된 이 InDel 마커들의 경우 변이들이 한국 품종과 그 교배친에서 안정적으로 유지되어 추가 마커의 개발 없이도 새로 육성되는 품종의 판별에도 적용될 수 있다고 하였다(Sohn et al., 2017a). 또한 202개 InDel 마커를 품종간 유연관계 분석과 염색체 재조합 양상의 구명에도 적용하여 이들 마커의 육종적 활용 가능성도 제시하였다(Sohn et al., 2017b).
본 연구에서는 Sohn et al. (2017a)이 국내 콩 149개 품종 판별에 적용할 수 있다고 선발한 27개 InDel 마커를 이후 개발된 신품종 20개에 적용하여 품종판별용 마커로서 범용성을 검증하고 신품종의 구별성과 균일성을 확인하여 품종보호를 위한 기초 자료를 확보하고자 하였다. 또한 신품종을 포함한 국내 품종의 유전적 다양성을 분석하여 향후 변이 확대를 위한 유전자원 도입과 원하는 영역의 선발을 위한 교배조합 예측 등 육종 효율을 개선하기 위해 27개 InDel 마커의 활용 가능성을 확인하고자 하였다. 본 연구에 사용된 InDel 마커는 품종 외에도 농업유전자원센터(http://genebank.rda.go.kr)에 보유되어 있는 수만 여점의 콩 유전자원에 적용하여 육종소재는 물론 화장품, 기능성 식품 등 부가가치가 높은 산업화 자원을 개발하기 위한 유전적 다양성분석의 기반기술로 활용될 수 있을 것으로 기대한다.
재료 및 방법
시험재료 및 DNA 추출
시험재료로 국립식량과학원 남부작물부(19품종)와 경기도농업기술원(‘연풍’)에서 2012년도 이후 육성된 20개 품종을 분양받아 사용하였다(Table 1). 용도 별로 장류 및 두부용 9품종, 밥밑용 5품종, 나물용 4품종, 단기성이 2품종에 해당된다. 시험재료는 완전임의배치법으로 온실에서 제 1본엽이 전개되는 단계에서 어린잎을 품종마다 5개체씩 채취하였다. Genomic DNA는 대량 DNA 추출 장치 Biosprint96 (Qiagen, Hilden, Germany)을 이용하여 Biosprint96 DNA plant kit (Qiagen, Hilden, Germany)를 이용하여 96 시료 단위로 개체별 DNA를 추출하였다. DNA 농도는 분광광도계(NanoVue Plus, Bio Chrom, USA)를 이용하여 측정하였으며 PCR 실험을 위하여 최종 농도 20 ng/μL로 희석하고 바코딩에는 품종당 5개체 DNA를 혼합하여 사용하였다.
Table 1. List of 20 soybean new cultivars used in this study
|
Utilization
|
Cultivar
|
Registered Year
|
Cross combination
|
|
Bean sprout
|
Aram
|
2019
|
Bosug/Camp
|
|
Haepum
|
2015
|
Bosug/Suwon214ho
|
|
Haewon
|
2017
|
Bosug/Somyeong
|
|
Soyeon
|
(2017)z |
Sokang/Nogchae
|
|
Cooking with rice
|
Cheongja4ho
|
2018
|
Milyang 148ho/Daemang
|
|
Cheongja5ho
|
2019
|
Milyang181ho (IT269976)/YS1886
|
|
Cheongmiin
|
(2014)z |
Cheongja/Daemang
|
|
Socheongja
|
2019
|
Suwon214ho/YCL14
|
|
Taecheong
|
2018
|
Milyang136ho/Danpahuekdaedoo//Cheongja3ho
|
|
Soy sauce & Tofu
|
Daechan
|
2017
|
Suwon224ho/Dongsan121ho
|
|
Daepung2ho
|
2017
|
Daepung/Suwon190ho//Daewon
|
|
Jinpung
|
2015
|
Daepung/SS01211
|
|
Miso
|
2019
|
Daemang/Jinpumkong2ho
|
|
Pyeongwon
|
2019
|
SS98205/Daemang2ho (IT 216351)
|
|
Saegeum
|
2016
|
Daepung/Suwon 190ho//Daewon
|
|
Seonpung
|
2016
|
Suwon224ho/Dongsan121ho
|
|
Taeseon
|
2017
|
Shingi/Taekwang
|
|
Yongpoong
|
2012
|
Suwon191ho/Suwon196ho
|
|
Early maturity
|
Duruol
|
2017
|
Hwaoemputkong/Suwon191ho
|
|
Jangol
|
2017
|
SI993773/Suwon192ho
|
zTwo cultivars, Soyeon and Cheongmiin were applied for the protection of new varieties of plants at the year in parenthesis, but are not registered yet.
InDel 마커의 PCR 분석 및 바코드화
본 실험에서는 Sohn et al. (2017a)이 202개 InDel 마커 중 품종판별용으로 선발한 27개 마커를 이용하였는데 그 중 Sindel_ 12_02마커는 비특이적인 PCR 증폭으로 인해 Sindel_12_11로 대체하여 사용하였다(Table 2). Sohn et al. (2017b)이 제시한 방법에 따라 20 μL 반응액[20 ng genomic DNA, 2 pmol primer, 1X DNA PCR premix (Genetbio, Korea)]으로 PCR (Biometra, Germany)을 수행하였다. PCR 조건은 95℃에서 5분 간 초기변성하고, 95℃에서 30초 간 변성단계를 거쳐, 55℃에서 30초 간 결합, 그리고 75℃에서 30초 간 신장단계를 35회 반복한 후, 최종적으로 72℃에서 5 분간 신장하였다. 먼저 1% 아가로스겔에서 PCR 증폭 여부를 확인하고, 증폭이 된 경우 3% 아가로스겔 대신 정밀한 DNA 사이즈 비교를 위해 capillary 방식의 LabChip (PerkinElmer, Massachusetts, USA)을 이용하여 자동 전기영동을 수행하였다.
Table 2. Details of 27 InDel markers used for the discrimination of 20 new soybean cultivars in this studyz
|
No.
|
Marker
|
Chromosome
|
Position
(bp)
|
Forward sequence
|
Reverse sequence
|
Amplicon
size (bp)
|
Annealing
Tm (℃)
|
|
1
|
Sindel_01_19
|
1
|
49,364,344
|
AACTAATTTTTGCACATCCC
|
AATGGAAAATGTTGGCTAGA
|
109
|
58
|
|
2
|
Sindel_02_13
|
2
|
9,295,181
|
GGTACAAGAATGAGTGGGTG
|
TGACACATTGACACTCATTAAA
|
91
|
58
|
|
3
|
Sindel_03_13
|
3
|
6,168,100
|
AAACATGAATGGTGCTTTTT
|
TAAAGCCCCAAAAGTGAATA
|
110
|
58
|
|
4
|
Sindel_03_19
|
3
|
45,101,996
|
ACTTGCCAGTTTGGTGTAAG
|
CCACGAGTACTGAGTGCATA
|
101
|
60
|
|
5
|
Sindel_03_20
|
3
|
47,630,105
|
GACTGTATTTGTAAAGTAGGAGTTT
|
GCCTTTTACTTTTCATCCTTC
|
116
|
58
|
|
6
|
Sindel_05_01
|
5
|
269,251
|
TGCTTAGAGTTTGGTTTGGT
|
CCGACACATCTTCTCCTCTA
|
84
|
58
|
|
7
|
Sindel_07_04
|
7
|
15,115,508
|
ATGCTAAAAATTGGATTTGC
|
CAGAAGAAGAAAGAAAGCACA
|
104
|
58
|
|
8
|
Sindel_07_18
|
7
|
39,130,876
|
CAAATTAATGTTTCGAAAAGC
|
AGAATTAGGTCTGTGGGACC
|
119
|
60
|
|
9
|
Sindel_08_03
|
8
|
2,765,869
|
ACCAATATCGATCTTTACCAA
|
TCACTCTCTTAGCATATTAATCAAA
|
112
|
60
|
|
10
|
Sindel_09_05
|
9
|
17,473,246
|
TCCGATCAGTCTCTAACACC
|
GCAAGAAAATTGAATTGGAG
|
113
|
59
|
|
11
|
Sindel_10_13
|
10
|
13,339,511
|
CTTTTACAATATTGAGGCCA
|
TTTAAATTGAAATTCCCCAA
|
96
|
55
|
|
12
|
Sindel_11_04
|
11
|
8,086,867
|
TTTGCATGTTTTGTATTTGG
|
AACAAATTAAAGAGAAGGCG
|
94
|
58
|
|
13
|
Sindel_11_09
|
11
|
38,153,075
|
CCCACACGGTTTTAATATGT
|
GACATTTGATTTCGTTTGTCT
|
117
|
58
|
|
14
|
Sindel_12_11y |
12
|
613,111
|
CCATGTCTCCTACAATGCAA
|
TGCACACAAGAAAAGAATCA
|
119
|
58
|
|
15
|
Sindel_12_18
|
12
|
33,886,457
|
TTGAAAATAGGGAAACCTGA
|
AACTGATGTTAGACAAGCCC
|
115
|
60
|
|
16
|
Sindel_13_09
|
13
|
38,116,607
|
ACATGACATTTAAGGGATGC
|
TTTCACTCTTTAGGTGCCTT
|
117
|
58
|
|
17
|
Sindel_13_18
|
13
|
25,633,692
|
GATCAAATCACTAAAATGTAGTCC
|
TCCAGATAAGAAAACAAAAACA
|
90
|
58
|
|
18
|
Sindel_14_04
|
14
|
7,146,867
|
TGTCAACCAATCTTTGTATAACAT
|
TCCTATACACGACGACTTCC
|
113
|
60
|
|
19
|
Sindel_14_15
|
14
|
22,505,689
|
TCACATACTATAGCATCCCGT
|
CGCTAGAGGTTGAAGATGAT
|
98
|
60
|
|
20
|
Sindel_15_12
|
15
|
5,031,193
|
TTTGAGGGTGTTTCTCACTT
|
TCAAAATTCAGTTATGTCCAA
|
98
|
58
|
|
21
|
Sindel_16_03
|
16
|
5,597,875
|
TCGGTCATTGGTATTTGATT
|
TGTGTAATTGTGTTATTGAATTAT
|
119
|
58
|
|
22
|
Sindel_17_05
|
17
|
15,720,082
|
TCCTCCTTTTGAAATGTGTC
|
CAAATCAAATGGAAGAAATACA
|
115
|
58
|
|
23
|
Sindel_18_16
|
18
|
39,438,923
|
TGCATGTGTCTGCATCTTAT
|
ACCATAACCCAAAATGACCT
|
82
|
60
|
|
24
|
Sindel_19_16
|
19
|
35,292,541
|
AGCAACTGCAAAAATCCTTA
|
TTTTCTGTATCTGTATGTTGTTC
|
98
|
60
|
|
25
|
Sindel_19_17
|
19
|
37,409,060
|
TGTCTGTGTGTTGAATGGAG
|
ACAACCTGTCAAACAAGTCC
|
90
|
58
|
|
26
|
Sindel_20_07
|
20
|
34,305,586
|
GCTAAGCAATTAGCAACTCAA
|
TTTATAATGCATGCACATTTTT
|
119
|
58
|
|
27
|
Sindel_20_09
|
20
|
39,540,342
|
GCTTTGATTTGGTCATGTTT
|
GTTTGTCGCAAAAGGTTAAG
|
102
|
60
|
zDetails of 27 markers were extracted among information of a total of 202 InDel markers developed by
Sohn et al. (2017a).
yThe marker Sindel_12_02 selected for cultivar identification analysis by
Sohn et al. (2017a) is replaced to Sindel_12_11 located on the same chromosome Gm19 because of non-specific PCR products.
27개 InDel 마커에 대한 증폭산물은 마커별 콩 표준유전체인 Williams 82 품종의 증폭산물과 같은 크기이면 ‘a’로 다른 크기이면 ‘b’로 판정하였다. 이 정보를 품종인식 바코드 시스템(MyCrops_Soybean)에 입력하여 ‘0’과 ‘1’의 디지털 신호로 변환하고 ‘0’은 흰색, ‘1’은 검은색의 바코드로 표시하여 가로는 마커의 염색체 번호로 세로는 마커의 염색체별 물리적 위치로 하여 2차원 바코드로 전환하였다(Sohn et al., 2017a).
유전적 유사도 및 유연관계분석
본 실험에서 바코딩한 20개 신품종과 기존 품종 간에 유사도를 비교하기 위해 콩 품종인식 바코드 시스템에 포함되어 있는 149 품종에 대한 27개 InDel 마커의 유전형을 추출하여 유전적 유사도 분석에 사용하였다. 유전적 유사도는 비교하는 두 품종 사이에 27개 마커별 같은 유전형을 가진 마커수를 백분률(두 품종 사이 동일한 유전형의 개수/27×100)로 표시하였다.
품종 간 유연관계는 R 통계프로그램(3.5.1 버전)에서 “dist” 함수(binary 옵션)와 “hclust” 함수(ward.D 옵션)를 이용하여 계층적 병합 군집분석을 수행한 뒤 시각화는 “plot.hclust” 함수를 사용하여 dendrogram을 작성하였다(The R Project for Statistical Computing R version 3.5.1 from https://www.r-project.org).
계보도 작성
콩 품종 20개에 대한 교배모본 정보를 활용하여 교배모본 선대의 정보를 수집하였다. Lee et al. (2015)이 콩 품종 계보에 대해 보고한 논문, 농촌진흥청 농사로(http://www.nongsaro.go.kr), 2018 주요 식량 밭작물 품종해설서(국립식량과학원, 2019) 등에서 정보를 수집하여 동일한 품종 및 계통을 묶어서 계보도를 작성하였다.
PIC (polymorphism information contents) 계산
본 실험에서 바코딩한 20개 신품종의 바코딩 정보와 콩 품종인식 바코드 시스템에 포함되어 있는 149품종에 대한 27개 마커별 유전형 정보를 이용하여 마커별 유전적 다형성(PIC)을 분석하였다. 계산은 Anderson et al. (1993)이 제시한 방법에 따라 R 통계프로그램(3.5.1 버전)에서 “table” 함수와 “lapply” 함수를 이용하여 PIC를 계산하였다.
결과 및 고찰
콩 27개 InDel 마커를 이용한 신품종 구분과 유사도 분석
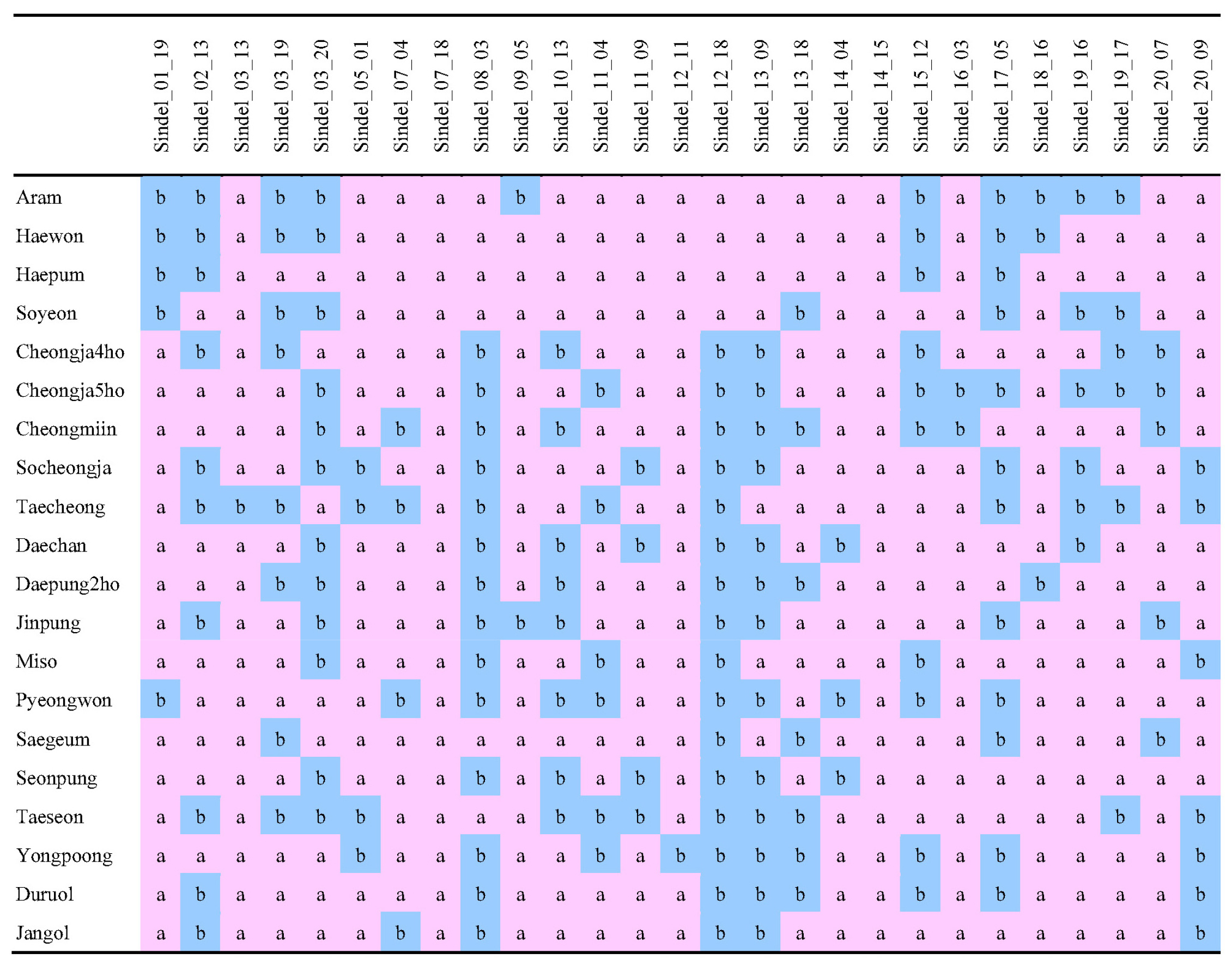
2012년도 이후 다양한 용도로 개발된 콩 신품종 20개(장류 및 두부용 9품종, 밥밑용 5품종, 나물용 4품종, 단기성 2품종)의 유전적 다양성 분석 및 품종 구분을 위해 Sohn et al. (2017a)이 제시한 27개 InDel 마커를 이용하여 신품종의 유전형을 분석하였다(Supplementary Fig. 1). 품종인식 바코드 시스템(MyCrops_Soybean)에서 유전형 정보를 디지털화하여 2차원 바코드로 전환하였으며(Fig. 1), 품종인식 시스템의 149품종과 비교하여 품종 간 유사도를 계산하였다(Supplementary Table 1). 20개 신품종과 149개 품종과의 유사도는 평균 61.3%이고, 최저 25.9%에서 최대 96.3%의 유사도로 완전 일치(100%)되는 바코드는 없어 20개 신품종의 유전적 구별성을 모두 확인할 수 있었다.

Fig. 1.
2-Dimensional barcoding representation of the polymorphisms revealed by 27 InDel markers in the 20 soybean cultivars. The white square located in the position represented same allele with Williams 82’s and black square represented different allele with Williams 82’s. Horizontal axis symbolized chromosome number of soybean and vertical axis exhibited the position of markers on each chromosome.
가장 낮은 유사도(25.9%)는 나물콩 용도의 신품종 ‘아람’(농촌진흥청 육성)과 품종인식 바코드 시스템에 포함된 두부 및 장류콩인 ‘기찬’(강원도농업기술원 육성) 사이에서 나타났다. 가장 높은 유전적 유사도(96.3%)는 두 신품종인 ‘대찬’ 과 ‘선풍’ 사이로, 두 품종의 바코드를 비교하여 27개 마커 중 Sindel_19_ 16 마커에서만 차이를 발견하였다(Fig. 2A). ‘대찬’ 과 ‘선풍’은 동일한 교배 모부본(‘수원224호’/’동산121호’)에서 유래되어 고세대 때 분리 선발된 품종들로 27개 InDel 마커로 명확하게 구분할 수 있음을 확인하였다(Fig. 2B).
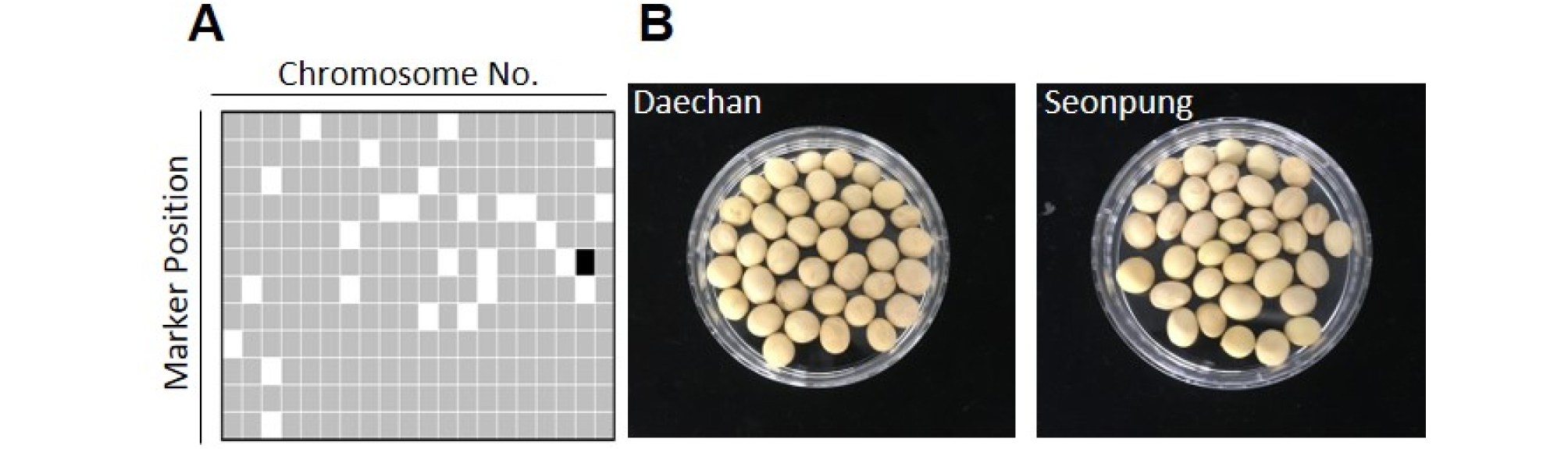
Fig. 2.
Comparison of 2-Dimensional barcode patterns between ‘Daechan’ and ‘Seonpung’ bred from cross of same parent lines, ‘Suwon224ho’ and Dongsan121ho’. (A) Homology comparison of 2-Dimensional barcode between ‘Daechan’ and ‘Seonpung’. Horizontal axis symbolized the number of chromosome where the 27 markers are located and vertical axis exhibited the position of markers on each chromosome. The white square represented the same between Daechan’s and Seonpung’s alleles, the black square represented a different allele between two cultivars’ alleles and the gray are null with no marker. (B) Seed morphology of ‘Daechan’ and ‘Seonpung’.
20개 신품종의 용도별 유전적 유사도를 비교해볼 때 나물콩 품종(4개)간 유전적 유사도는 평균 81.5% (74.1~88.9%)로 두부 및 장류콩 품종(9개)간 평균 유사도 67.9% (48.1~96.3%)와 밥밑용 품종(5개)간 평균 유사도 63.0% (40.7~74.1%)에 비해 크게 높아 나물콩 용도의 신품종 간의 유전적 다양성이 낮음을 알 수 있었다. 이러한 결과는 국내 나물콩이 소립종 선발을 주요 육종목표로 하고 있어 교배 모본의 풀이 제한적으로 사용되고 있다는 Lee et al. (2015)의 보고와도 일치한다.
유연관계분석
품종별 27개 InDel 마커의 유전형 정보를 이용하여 신품종 20개와 품종인식 바코드 시스템의 149품종에 대한 유연관계분석을 수행하였다(Fig. 3). 크게 두 개의 그룹으로 분류되었는데 I 그룹에는 ‘태청’부터 ‘석량풋콩’까지 103개 품종이, II 그룹에는 ‘보석’부터 ‘새금’까지 66개 품종이 각각 포함되었다. I 그룹에는 장류 및 두부콩이 49.5% (51품종)로, II 그룹에는 나물콩이 53.0% (35품종)로 우점하는 것으로 나타났다(Fig. 4).
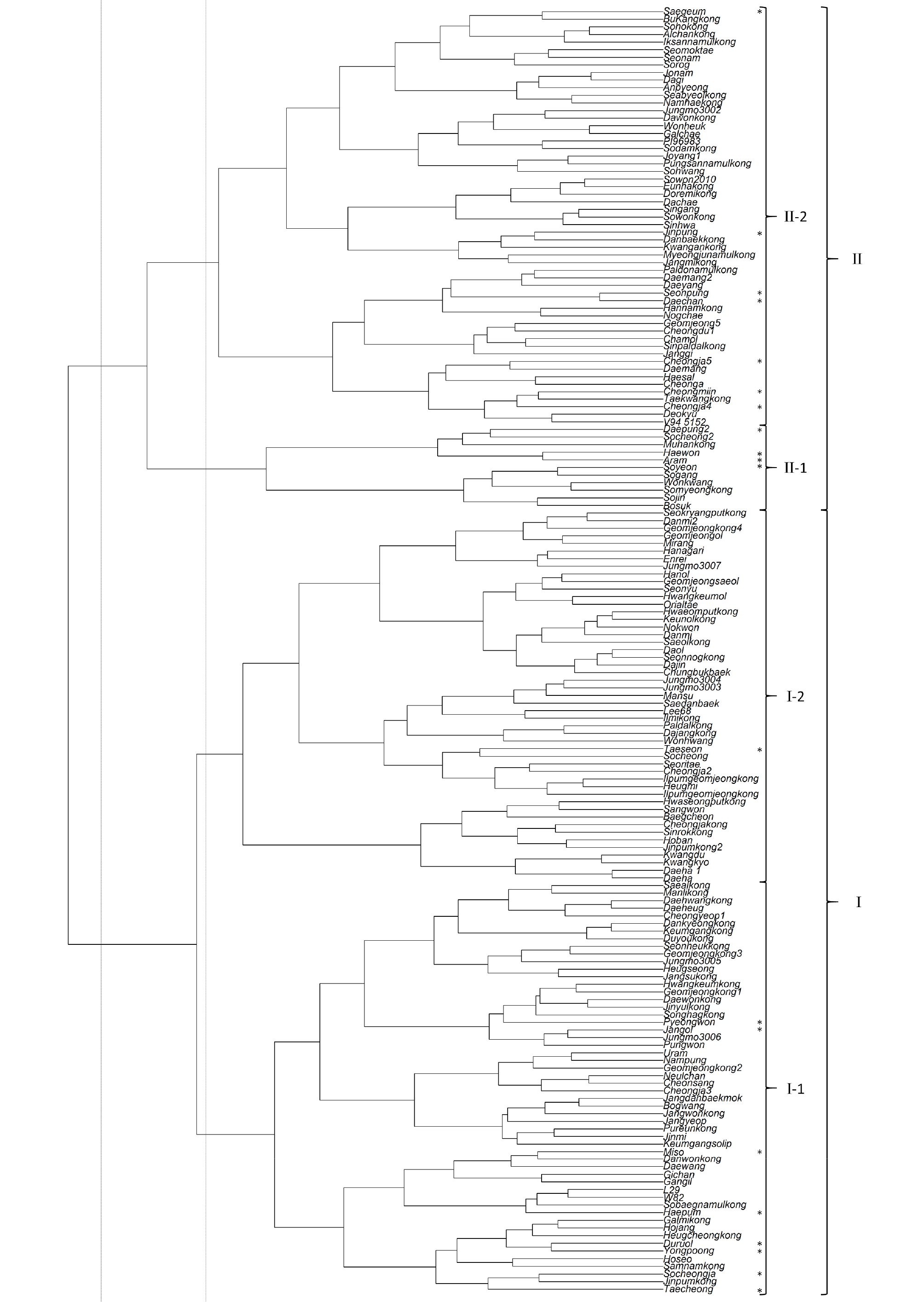
Fig. 3.
Hierarchical clustering tree of 169 soybean cultivars based on 27 InDel markers. Cultivars with asterisks are 20 new soybean cultivars.
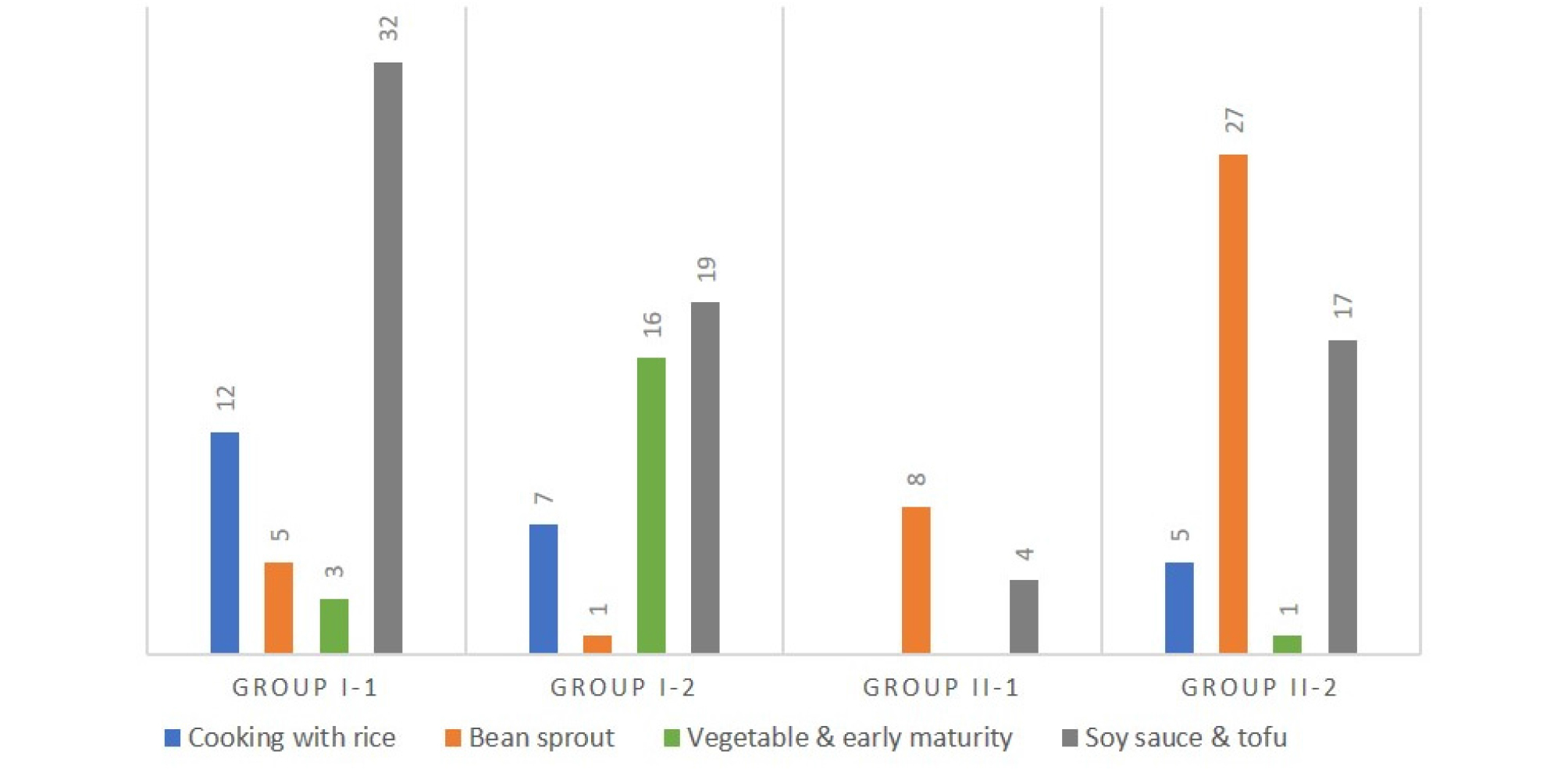
Fig. 4.
Number of cultivars by soybean utilization types in four subgroups based on hierarchical clustering result. Twelve cultivars of unknown utilization type were excluded.
용도별로 그룹 구성을 보면 밥밑콩(24품종)의 경우 79.2% (19품종)가 I 그룹에 포함되었으며, I-1 소그룹과 I-2소그룹에 각각 50.0% (12품종), 29.2% (7품종)이 포함되었다(Fig. 4). 나물콩(41품종)의 경우 85.4% (35품종)가 II 그룹에 분포하였고, 풋콩 및 단기성(20품종)은 95.0% (19품종)가 I 그룹, 특히 I-2 그룹에 80.0% (16품종)가 분포하였다(Fig. 4). 이러한 결과는 Sohn et al. (2017b)의 202개 InDel마커를 활용한 국내품종 유연관계 및 유전구조분석에서 유전집단이 우리나라 콩 육종프로그램에 따라 구분된 네 가지의 용도와 연관성이 높다는 보고와 일치한다. 특히, 본 연구에서는 변이밀집영역(dVB)으로부터 개발된 202개 마커 전체를 사용하는 대신 품종구분용으로 선발된 27개 마커만으로도 풋콩 및 단기성 콩의 80%가 I-2그룹에 속하고 나물콩의 65.9%가 II-2 그룹에 속하는 등 이들의 유전적 다양성이 제한적인라는 것을 군집분석을 통해 확인할 수 있었다.
장류 및 두부콩(72 품종)의 경우는 I-1, I-2, II-2 소그룹에 각각 44.4% (32 품종), 26.4% (19 품종), 23.6% (17품종)씩 포함되어 있으며 나물콩, 풋콩 및 단기성 품종들에 비해 상대적으로 소그룹별 고르게 분포하는 것을 알 수 있었다. 이러한 분포는 장류 및 두부콩의 오랜 육종 과정에서 다양한 그룹 내 재래종 또는 품종의 유전자 조합(genetic reshuffling)을 지속적으로 유도한 결과로 예측하고 있다(Sohn et al., 2017b).
신품종 계보도에 따른 유연관계 분석
20개 신품종의 계보도는 크게 두 그룹으로 나누어지며 밭밑콩 ‘청자5호’와 ‘태청’, 단기성인 ‘장올’은 두 그룹에 속하지 않고 독립적으로 존재하였다(Fig. 5). A 그룹은 외래 유전자원(Hill, D69-7816, Camp)을 도입하여 ‘은하콩’, ‘남해콩’ 등으로 이어지는 나물콩 품종육성 주요 계보로서 나물콩류 신품종 4개(‘아람’, ‘소연’, ‘해원’, ‘해품’)가 모두 포함되어 있다. B 그룹은 ‘신팔달 2호’로부터 내려오는 장류 및 두부콩류의 신품종 8품종(‘대찬’, ‘대풍’, ‘미소’, ‘새금’, ‘선풍’, ‘진품’, ‘태선’, ‘평원’)이 주로 포함되어 있다.
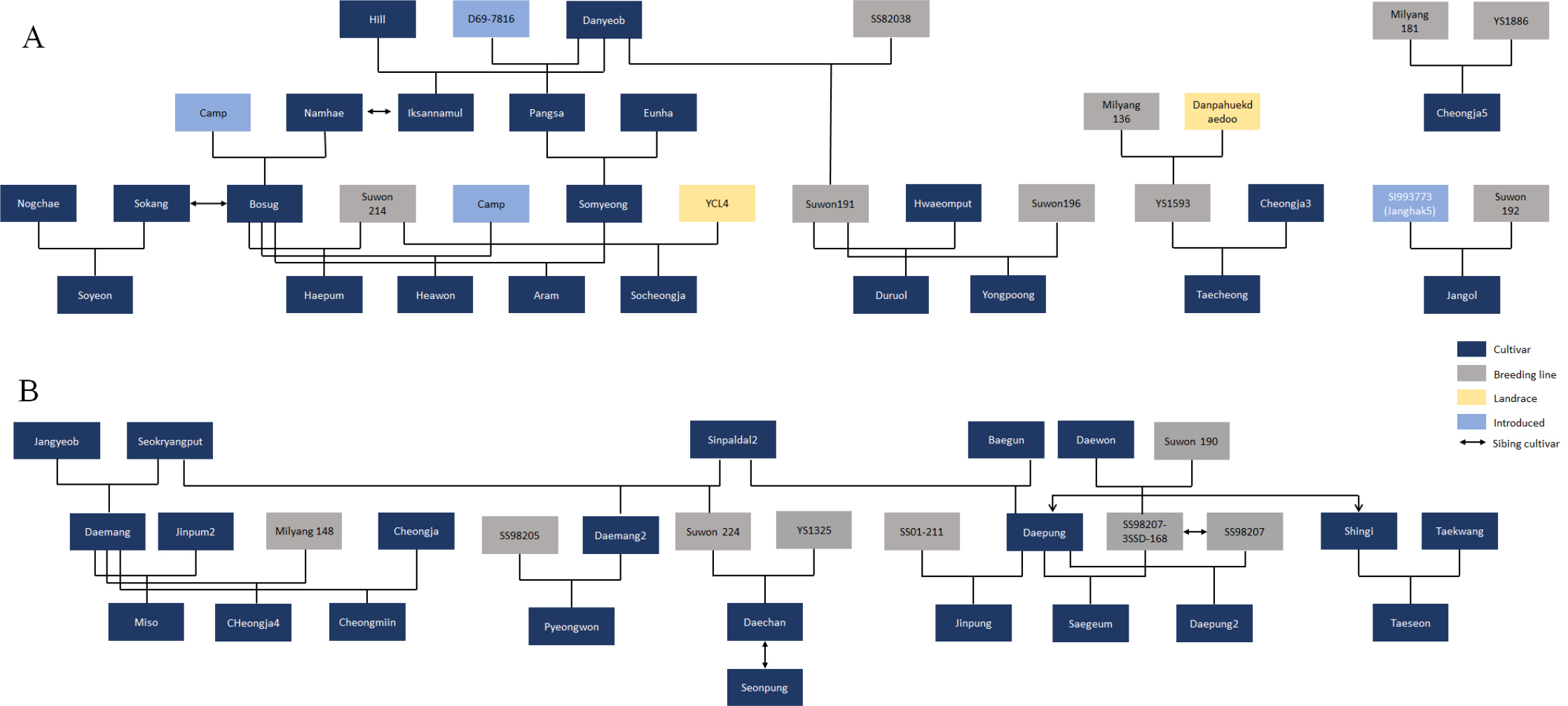
Fig. 5.
Reconstruction pedigrees of 20 new soybean cultivars. The pedigrees were roughly divided two groups. One group mainly consisted of Bean sprout usage and the other group mainly consisted of Soy sauce and Cooking with rice usage. ‘TaeCheong’, ‘Jangol’ and ‘Cheongja5’ were not integrated because of no linked or shared parent lines.
나물콩류 신품종인 ‘아람’, ‘소연’, ‘해원’, ‘해품’의 교배친으로 ‘보석’ 또는 ‘보석’의 자매친인 ‘소강’이 공통적으로 이용되었는데, 군집분석에서는 ‘아람’, ‘소연’, ‘해원’은 나물콩이 주로 포함된 II-1 소집단에 그들의 교배친인 ‘보석’, ‘소강’, ‘소명’ 등과 나란히 위치하였으나 ‘해품’의 경우 또 다른 교배친인 ‘수원 214호’를 공유하는 밭밑용 ‘소청자’와 함께 I-1 소집단 내 포함되었다(Fig. 3). ‘신팔달2호’에서 유래되어 동일한 교배친(‘수원224호’/’동산121호’)을 가지는 ‘대찬’과 ‘선풍’, ‘수원 191호’를 한쪽 교배친으로 공유하는 ‘두루올’과 ‘연풍’, ‘대망’을 교배친으로 공유하는 ‘청자4호’와 ‘청미인’은 군집분석에서 각각 서로 인접하게 위치하였다(Fig. 3). 따라서 27개 InDel 마커에 의한 유연관계 분석은 품종의 계보도와 교배모부본의 정보를 잘 반영하고 있음을 확인할 수 있었다.
품종판별
콩 신품종 20개와 품종인식 바코드 시스템에 있는 149품종을 판별하기 위한 최소 InDel 마커를 두 단계 과정을 통해 선발하였다. 1단계에서는 27개 마커 중 PIC 값이 높은 마커를 선정하였고, 2단계에서는 1단계에서 선발된 마커로 구별되지 않은 나머지 품종을 구분하기 위해 품종별로 구분 마커를 추가 선정하였다(Table 3). 1단계에는 신품종 20개와 품종인식 바코드 시스템에 포함된 149개 품종을 모두 합한 169품종에 대한 27개 마커의 PIC 값을 분석하여 높은 순으로 7개 마커를 선정(평균 0.371)하였으며, PIC 값(0.369)이 높지만 신품종 간에 다형성을 보이지 않는 Sindel_07_18 마커는 제외하였다. 1단계 선발된 7개 마커를 이용하면 신품종 중 7개 품종 ‘두루올’, ‘미소’, ‘새금’, ‘청미인’, ‘청자5호’, ‘태선’, ‘평원’을 품종인식 바코드 시스템에 있는 모든 품종과 구분할 수 있다. 1단계 과정을 통해 구분되지 않는 신품종 13개를 구분하기 위해 2단계에서는 품종별 구분 마커를 선정하였는데 ‘해품’의 경우 1단계 선정마커 7개를 이용했을 때 기존 149품종 대부분과 구분되었으나 ‘신화’ 품종과는 차이가 없어 이를 구분하기 위해 Sindel_20_09 마커를 추가 선정하였다. ‘아람’ 품종의 경우 1단계 마커를 이용하면 신품종을 포함한 168품종 중 3개 품종(‘소록’, ‘진품’, ‘해원’)과 구분되지 않아 이들을 구분하기 위해 Sindel_03_19와 Sindel_19_16 마커를 추가 선정하였다. 따라서 본 연구의 신품종 20 개를 기존 품종과 판별하기 위한 최소 마커는 품종에 따라 최소 7개에서 최대 9개까지 선정될 수 있었다. 집단구조, 유전적 다양성 및 유연관계 분석 등에는 유전체 전체를 대변할 수 있는 다수의 마커가 활용되어야 하나 품종 진위를 판별하기 위해서는 반드시 모든 마커를 사용할 필요없이 품종에 따라 마커 수를 최소화할 수 있다.
Table 3. Changes of PIC value of 27 markers and markers selected for new cultivars identification
|
Markers
|
PIC
| |
Cultivar identification
|
|
Total 169
cultivars
|
149 cultivars
(A)
|
20 cultivars
(B)
|
Changes
(B-A)
|
1st step
|
2nd step
|
|
Sindel_01_19
|
0.3299
|
0.3327
|
0.3047
|
-0.0280
| | |
Jangol, Seonpung
|
|
Sindel_02_13
|
0.3730
|
0.3725
|
0.3750
|
0.0025
| |
20 cultivars
| |
|
Sindel_03_13
|
0.2835
|
0.2993
|
0.0905
|
-0.2088
| | | |
|
Sindel_03_19
|
0.3471
|
0.3440
|
0.3648
|
0.0208
| | |
Aram, Cheonja4ho,
Daepung2ho, Socheongja
|
|
Sindel_03_20
|
0.3735
|
0.3741
|
0.3648
|
-0.0093
| |
20 cultivars
| |
|
Sindel_05_01
|
0.2494
|
0.2466
|
0.2688
|
0.0222
| | | |
|
Sindel_07_04
|
0.3442
|
0.3505
|
0.2688
|
-0.0817
| | |
Haewon
|
|
Sindel_07_18
|
0.3685
|
0.3744
|
0.0000
|
-0.3744
| | | |
|
Sindel_08_03
|
0.3074
|
0.3036
|
0.3318
|
0.0282
| | | |
|
Sindel_09_05
|
0.1797
|
0.1817
|
0.1638
|
-0.0179
| | | |
|
Sindel_10_13
|
0.3000
|
0.2854
|
0.3648
|
0.0794
| | | |
|
Sindel_11_04
|
0.3664
|
0.3690
|
0.3318
|
-0.0372
| |
20 cultivars
| |
|
Sindel_11_09
|
0.2439
|
0.2403
|
0.2688
|
0.0285
| | | |
|
Sindel_12_11
|
0.3613
|
0.3357
|
0.0905
|
-0.2452
| |
20 cultivars
| |
|
Sindel_12_18
|
0.2494
|
0.2466
|
0.2688
|
0.0222
| | | |
|
Sindel_13_09
|
0.3270
|
0.3229
|
0.3515
|
0.0286
| | | |
|
Sindel_13_18
|
0.3744
|
0.3750
|
0.3515
|
-0.0235
| |
20 cultivars
| |
|
Sindel_14_04
|
0.2878
|
0.2948
|
0.2225
|
-0.0724
| | | |
|
Sindel_14_15
|
0.2158
|
0.2359
|
0.0000
|
-0.2359
| | | |
|
Sindel_15_12
|
0.3748
|
0.3747
|
0.3750
|
0.0003
| |
20 cultivars
| |
|
Sindel_16_03
|
0.2878
|
0.2993
|
0.1638
|
-0.1355
| | | |
|
Sindel_17_05
|
0.3746
|
0.3736
|
0.3648
|
-0.0088
| |
20 cultivars
| |
|
Sindel_18_16
|
0.0558
|
0.0261
|
0.2225
|
0.1964
| | | |
|
Sindel_19_16
|
0.2698
|
0.2586
|
0.3318
|
0.0732
| | |
Aram, Daechan, Haewon,
Jinpung, Soyeon,
Taecheong, Yongpoong
|
|
Sindel_19_17
|
0.2745
|
0.2644
|
0.3318
|
0.0674
| | | |
|
Sindel_20_07
|
0.1940
|
0.1733
|
0.3047
|
0.1314
| | | |
|
Sindel_20_09
|
0.3324
|
0.3292
|
0.3515
|
0.0223
| | |
Haepum
|
| Average |
0.2980
|
0.2957
|
0.2677
|
-0.0280
| | | |
마커별 PIC 변화
27개 InDel 마커의 다형성 변화를 확인하기 위해 품종인식 바코드 시스템 내 149품종과 신품종 20개에 대한 마커별 PIC 값의 변화를 확인하였다(Table 3). 신품종 20개에 대한 PIC 결과는 평균 0.2677로, Sohn et al. (2017a)이 149개 품종에 바코딩한 결과의 평균 PIC인 0.2957에 비해 소폭(0.0280) 감소하였는데 이는 149품종에 비해 신품종에서 다양성이 매우 낮아지는 일부 마커(Sinde1_03_13, Sindel_07_18, Sindel_12_11, Sindel_14_15)의 영향때문이다. 27개 InDel 마커 중 14개 마커는 신품종에서 PIC 값이 소폭 증가하였으며 특히 Sindel_18_16 마커의 경우 0.1964 증가하였다. 종합적으로 보면 27개 마커 중 다형성이 크게 감소(4종) 또는 증가(1종)하는 일부 마커를 제외하고 나머지 마커들은 PIC의 큰 변화가 관찰되지 않았다.
이러한 이유는 27개 InDel 마커가 dVB에서 유래되어 변이가 국내 품종에서 안정적으로 유지되기 때문이며 특히 202개 InDel 마커 중에서 품종판별용으로 선발된 27개 마커의 다형성이 상대적으로 높기 때문이다. InDel 마커가 유래된 dVB는 표준유전체(‘Williams 82’)와 한국 품종(‘백운’ 등 6품종) 사이 단일염기서열변이(SNV)가 10 Kb 당 4개 이상 존재하는 영역으로, 특히 콩의 평균 LD (linkage disequilibrium) 블록 90~574 Kb 보다 짧기 때문에 품종육성 과정에서 유전자 재조합이 잘 일어나지 않아 변이들이 후대에 안정적으로 유전되는 특징이 있다(Kim et al., 2014; Sohn et al., 2018).
본 실험에서는 dVB에서 유래한 27개 InDel 마커를 신품종에 적용하여 향후 새로 육성되는 콩 품종의 판별기술로서 InDel 마커의 범용성을 확인하였다. InDel 마커를 이용한 판별기술은 품종의 구별성과 균일성을 보다 정확하게 판단하는 기술로서 품종등록 시 육성가의 권리보호에 기여하며, 국산콩과 수입콩의 원산지 구분을 통해 국내 콩 생산자와 소비자 보호에도 기여할 수 있다. 이 외에도 27개 InDel 마커는 품종의 군집분석, 다양성 분석, 유전자 재조합(reshuffling) 양상 예측 등에도 이용되어 교배육종 과정 중 신규자원을 이용한 유전변이의 도입 및 확대, 목표형질의 선발 등 육종 효율 개선에도 도움이 될 수 있을 것으로 기대한다.
적 요
본 연구에서는 국내 콩 유전체의 변이밀집영역(dVB)에서 유래한 27개 InDel 마커를 신품종 20개에 적용하여 품종판별용 마커로서 범용성을 검증하고 신품종의 구별성과 국내 품종의 유전적 다양성을 확인하였다. 20개 신품종과 MyCrops에 포함된 기존 149개 품종과의 유사도는 평균 61.3%이고, 최저 25.9%에서 최대 96.3%의 유사도로 완전 일치(100%)되는 바코드는 없어 20개 신품종의 유전적 구별성을 모두 확인할 수 있었다. 유연관계를 분석한 결과에서는 신품종을 포함한 국내 169품종이 4개의 유전집단으로 구분되었으며 풋콩 및 단기성 콩의 80%가 I-2 소그룹, 나물콩의 65.9%가 II-2 소그룹에 주로 속한 반면, 장류 및 두부콩은 I-1 (44.4%), I-2 (26.4%), II-2 (23.6%) 소그룹에 고르게 분포하였다. 20개 신품종에 대한 계보도는 나물콩 주요 계보와 장류 및 두부콩으로 크게 두 그룹으로 나누어지며 유연관계분석을 뒷받침하였다. 품종판별을 위한 최소 마커를 선발하기 위해 PIC가 높은 공통마커와 품종별 특이마커를 선발하는 2단계 과정을 통해 품종에 따라 7~9개의 최소 마커로 신품종의 진위를 판별할 수 있었다. 이처럼 콩 변이밀집영역에서 유래된 27개 InDel 마커와 이를 이용한 신품종 바코드 정보의 지속적인 업데이트는 수입산에 대한 국산 품종의 보호와 육성가의 권리 증진에 기여하며, 더불어 육종과정 중 신규 유전변이를 도입하고 목표형질을 선발하는 등 육종 효율을 개선하는데도 도움이 될 것으로 기대한다.
Supplementary Fig. 1.
A diagram of genotypes of 20 soybean cultivars obtained using 27 InDel markers. The same allele with Williams 82’s one is represented by ‘a’ (pink) and the other by ‘b’ (blue).
Supplementary Table 1. Genetic similarity between soybean cultivars
|
Nokwon
|
Dajin
|
Danmi
|
Danmi2
|
Mirang
|
Sangwon
|
Seokryangputkong
|
Seonnogkong
|
Sinrokkong
|
Saeolkong
|
|
Pyeongwon
|
66.67
|
74.07
|
70.37
|
59.26
|
59.26
|
62.96
|
59.26
|
66.67
|
70.37
|
55.56
|
|
Cheongja5ho
|
70.37
|
70.37
|
74.07
|
77.78
|
70.37
|
59.26
|
77.78
|
62.96
|
59.26
|
59.26
|
|
Taecheong
|
44.44
|
51.85
|
55.56
|
51.85
|
51.85
|
55.56
|
44.44
|
59.26
|
55.56
|
55.56
|
|
Aram
|
44.44
|
44.44
|
55.56
|
44.44
|
44.44
|
40.74
|
44.44
|
44.44
|
40.74
|
40.74
|
|
Cheongja4ho
|
62.96
|
55.56
|
66.67
|
55.56
|
62.96
|
44.44
|
55.56
|
55.56
|
51.85
|
59.26
|
|
Soyeon
|
55.56
|
55.56
|
59.26
|
55.56
|
48.15
|
44.44
|
55.56
|
55.56
|
37.04
|
44.44
|
|
Daepung2ho
|
74.07
|
66.67
|
70.37
|
66.67
|
66.67
|
48.15
|
66.67
|
66.67
|
48.15
|
62.96
|
|
Daechan
|
66.67
|
59.26
|
62.96
|
66.67
|
59.26
|
55.56
|
66.67
|
59.26
|
55.56
|
55.56
|
|
Miso
|
81.48
|
74.07
|
77.78
|
81.48
|
81.48
|
55.56
|
74.07
|
66.67
|
70.37
|
70.37
|
|
Socheongja
|
59.26
|
59.26
|
70.37
|
66.67
|
66.67
|
62.96
|
59.26
|
66.67
|
62.96
|
62.96
|
|
Duruol
|
74.07
|
74.07
|
85.19
|
74.07
|
81.48
|
62.96
|
66.67
|
74.07
|
70.37
|
70.37
|
|
Haewon
|
55.56
|
55.56
|
66.67
|
48.15
|
55.56
|
44.44
|
48.15
|
55.56
|
51.85
|
51.85
|
|
Taeseon
|
59.26
|
51.85
|
62.96
|
59.26
|
66.67
|
48.15
|
51.85
|
59.26
|
48.15
|
62.96
|
|
Seonpung
|
70.37
|
62.96
|
66.67
|
62.96
|
62.96
|
51.85
|
62.96
|
62.96
|
59.26
|
59.26
|
|
Saegeum
|
62.96
|
62.96
|
66.67
|
55.56
|
55.56
|
44.44
|
55.56
|
62.96
|
44.44
|
51.85
|
|
Cheongmiin
|
74.07
|
74.07
|
70.37
|
74.07
|
74.07
|
55.56
|
74.07
|
66.67
|
55.56
|
62.96
|
|
Jangol
|
66.67
|
66.67
|
70.37
|
66.67
|
74.07
|
55.56
|
59.26
|
74.07
|
70.37
|
62.96
|
|
Jinpung
|
62.96
|
62.96
|
74.07
|
55.56
|
62.96
|
51.85
|
55.56
|
70.37
|
59.26
|
59.26
|
|
Haepum
|
59.26
|
59.26
|
70.37
|
51.85
|
59.26
|
48.15
|
51.85
|
59.26
|
55.56
|
55.56
|
|
Yongpoong
|
81.48
|
81.48
|
85.19
|
81.48
|
81.48
|
70.37
|
74.07
|
74.07
|
70.37
|
77.78
|
|
Keunol
kong
|
Hwaeom
putkong
|
Galchae
|
Geomjeongol
|
Geom
jeongkong
1
|
Geom
jeongkong
2
|
Geom
jeongkong
3
|
Geom
jeongkong
4
|
Daeheug
|
Seonheuk
kong
|
|
Pyeongwon
|
66.67
|
62.96
|
59.26
|
62.96
|
66.67
|
59.26
|
62.96
|
55.56
|
70.37
|
74.07
|
|
Cheongja5ho
|
70.37
|
66.67
|
62.96
|
66.67
|
55.56
|
55.56
|
74.07
|
74.07
|
59.26
|
62.96
|
|
Taecheong
|
51.85
|
48.15
|
51.85
|
48.15
|
59.26
|
66.67
|
55.56
|
55.56
|
48.15
|
59.26
|
|
Aram
|
51.85
|
48.15
|
44.44
|
33.33
|
44.44
|
59.26
|
48.15
|
48.15
|
48.15
|
44.44
|
|
Cheongja4ho
|
62.96
|
66.67
|
62.96
|
51.85
|
62.96
|
55.56
|
66.67
|
51.85
|
66.67
|
62.96
|
|
Soyeon
|
55.56
|
51.85
|
48.15
|
44.44
|
55.56
|
62.96
|
66.67
|
59.26
|
59.26
|
62.96
|
|
Daepung2ho
|
66.67
|
70.37
|
59.26
|
62.96
|
66.67
|
59.26
|
77.78
|
62.96
|
77.78
|
66.67
|
|
Daechan
|
59.26
|
62.96
|
66.67
|
62.96
|
66.67
|
51.85
|
70.37
|
70.37
|
70.37
|
59.26
|
|
Miso
|
74.07
|
77.78
|
59.26
|
77.78
|
74.07
|
74.07
|
77.78
|
77.78
|
70.37
|
66.67
|
|
Socheongja
|
66.67
|
62.96
|
74.07
|
55.56
|
66.67
|
59.26
|
62.96
|
70.37
|
55.56
|
51.85
|
|
Duruol
|
81.48
|
77.78
|
74.07
|
70.37
|
74.07
|
66.67
|
70.37
|
62.96
|
70.37
|
66.67
|
|
Haewon
|
62.96
|
59.26
|
55.56
|
44.44
|
55.56
|
70.37
|
51.85
|
51.85
|
59.26
|
48.15
|
|
Taeseon
|
59.26
|
62.96
|
51.85
|
55.56
|
51.85
|
44.44
|
70.37
|
62.96
|
55.56
|
59.26
|
|
Seonpung
|
62.96
|
66.67
|
70.37
|
66.67
|
70.37
|
55.56
|
74.07
|
66.67
|
74.07
|
62.96
|
|
Saegeum
|
62.96
|
59.26
|
62.96
|
51.85
|
62.96
|
62.96
|
66.67
|
59.26
|
66.67
|
62.96
|
|
Cheongmiin
|
66.67
|
70.37
|
59.26
|
70.37
|
66.67
|
59.26
|
70.37
|
62.96
|
77.78
|
66.67
|
|
Jangol
|
66.67
|
70.37
|
74.07
|
62.96
|
88.89
|
66.67
|
70.37
|
70.37
|
70.37
|
74.07
|
|
Jinpung
|
70.37
|
66.67
|
70.37
|
51.85
|
62.96
|
55.56
|
66.67
|
59.26
|
59.26
|
55.56
|
|
Haepum
|
66.67
|
62.96
|
66.67
|
48.15
|
66.67
|
66.67
|
55.56
|
55.56
|
55.56
|
59.26
|
|
Yongpoong
|
81.48
|
77.78
|
59.26
|
77.78
|
74.07
|
66.67
|
77.78
|
70.37
|
70.37
|
74.07
|
|
So
hwang
|
Ilpum
geomjeongkong
|
Jinyul
kong
|
Cheongja
kong
|
Cheongja2
|
Heugmi
|
Keumgang
kong
|
Nampung
|
Dajang
kong
|
Dankyeong
kong
|
|
Pyeongwon
|
51.85
|
59.26
|
81.48
|
59.26
|
55.56
|
62.96
|
70.37
|
62.96
|
70.37
|
74.07
|
|
Cheongja5ho
|
55.56
|
62.96
|
70.37
|
62.96
|
59.26
|
59.26
|
59.26
|
66.67
|
66.67
|
62.96
|
|
Taecheong
|
44.44
|
44.44
|
59.26
|
59.26
|
55.56
|
55.56
|
40.74
|
62.96
|
70.37
|
44.44
|
|
Aram
|
37.04
|
51.85
|
44.44
|
44.44
|
40.74
|
40.74
|
55.56
|
62.96
|
55.56
|
51.85
|
|
Cheongja4ho
|
55.56
|
77.78
|
70.37
|
62.96
|
66.67
|
74.07
|
59.26
|
66.67
|
81.48
|
55.56
|
|
Soyeon
|
40.74
|
55.56
|
48.15
|
40.74
|
44.44
|
44.44
|
66.67
|
66.67
|
51.85
|
62.96
|
|
Daepung2ho
|
51.85
|
74.07
|
66.67
|
51.85
|
55.56
|
62.96
|
70.37
|
70.37
|
62.96
|
66.67
|
|
Daechan
|
51.85
|
66.67
|
66.67
|
51.85
|
40.74
|
48.15
|
62.96
|
62.96
|
62.96
|
59.26
|
|
Miso
|
59.26
|
66.67
|
81.48
|
74.07
|
55.56
|
55.56
|
70.37
|
77.78
|
77.78
|
74.07
|
|
Socheongja
|
51.85
|
59.26
|
59.26
|
59.26
|
40.74
|
40.74
|
55.56
|
62.96
|
55.56
|
51.85
|
|
Duruol
|
51.85
|
74.07
|
74.07
|
66.67
|
62.96
|
62.96
|
70.37
|
70.37
|
70.37
|
66.67
|
|
Haewon
|
40.74
|
55.56
|
55.56
|
48.15
|
44.44
|
44.44
|
66.67
|
74.07
|
59.26
|
62.96
|
|
Taeseon
|
51.85
|
66.67
|
51.85
|
59.26
|
55.56
|
62.96
|
55.56
|
48.15
|
62.96
|
59.26
|
|
Seonpung
|
55.56
|
70.37
|
70.37
|
55.56
|
44.44
|
51.85
|
66.67
|
66.67
|
66.67
|
62.96
|
|
Saegeum
|
62.96
|
55.56
|
62.96
|
40.74
|
51.85
|
51.85
|
66.67
|
74.07
|
59.26
|
62.96
|
|
Cheongmiin
|
51.85
|
74.07
|
74.07
|
59.26
|
62.96
|
70.37
|
77.78
|
62.96
|
62.96
|
74.07
|
|
Jangol
|
59.26
|
66.67
|
81.48
|
74.07
|
62.96
|
62.96
|
70.37
|
62.96
|
70.37
|
66.67
|
|
Jinpung
|
62.96
|
70.37
|
62.96
|
55.56
|
44.44
|
51.85
|
59.26
|
66.67
|
66.67
|
55.56
|
|
Haepum
|
51.85
|
59.26
|
66.67
|
51.85
|
48.15
|
48.15
|
70.37
|
70.37
|
70.37
|
66.67
|
|
Yongpoong
|
66.67
|
66.67
|
81.48
|
66.67
|
62.96
|
62.96
|
70.37
|
70.37
|
62.96
|
74.07
|
|
Danbaek
kong
|
Danwon
kong
|
Daemang
|
Daemang2
|
Daeyang
|
Daewonkong
|
Daehwang
kong
|
Duyoukong
|
Manlikong
|
Mansu
|
|
Pyeongwon
|
62.96
|
74.07
|
55.56
|
66.67
|
66.67
|
81.48
|
74.07
|
74.07
|
55.56
|
66.67
|
|
Cheongja5ho
|
66.67
|
55.56
|
74.07
|
55.56
|
62.96
|
62.96
|
62.96
|
55.56
|
59.26
|
62.96
|
|
Taecheong
|
62.96
|
59.26
|
48.15
|
44.44
|
66.67
|
59.26
|
51.85
|
37.04
|
48.15
|
59.26
|
|
Aram
|
48.15
|
51.85
|
40.74
|
37.04
|
51.85
|
44.44
|
44.44
|
51.85
|
48.15
|
44.44
|
|
Cheongja4ho
|
66.67
|
55.56
|
59.26
|
55.56
|
48.15
|
62.96
|
62.96
|
55.56
|
66.67
|
62.96
|
|
Soyeon
|
44.44
|
62.96
|
51.85
|
55.56
|
62.96
|
55.56
|
55.56
|
62.96
|
66.67
|
62.96
|
|
Daepung2ho
|
55.56
|
59.26
|
62.96
|
74.07
|
59.26
|
66.67
|
74.07
|
66.67
|
85.19
|
74.07
|
|
Daechan
|
62.96
|
51.85
|
55.56
|
74.07
|
66.67
|
66.67
|
66.67
|
66.67
|
77.78
|
66.67
|
|
Miso
|
70.37
|
81.48
|
77.78
|
74.07
|
66.67
|
74.07
|
74.07
|
66.67
|
70.37
|
66.67
|
|
Socheongja
|
77.78
|
51.85
|
55.56
|
51.85
|
66.67
|
59.26
|
51.85
|
51.85
|
70.37
|
66.67
|
|
Duruol
|
77.78
|
74.07
|
62.96
|
59.26
|
66.67
|
66.67
|
66.67
|
66.67
|
70.37
|
81.48
|
|
Haewon
|
51.85
|
62.96
|
51.85
|
48.15
|
55.56
|
55.56
|
55.56
|
62.96
|
59.26
|
55.56
|
|
Taeseon
|
62.96
|
51.85
|
55.56
|
59.26
|
51.85
|
51.85
|
59.26
|
51.85
|
70.37
|
51.85
|
|
Seonpung
|
66.67
|
55.56
|
59.26
|
77.78
|
62.96
|
70.37
|
70.37
|
70.37
|
81.48
|
70.37
|
|
Saegeum
|
66.67
|
62.96
|
66.67
|
62.96
|
62.96
|
62.96
|
62.96
|
62.96
|
74.07
|
77.78
|
|
Cheongmiin
|
55.56
|
66.67
|
77.78
|
66.67
|
59.26
|
66.67
|
74.07
|
74.07
|
70.37
|
66.67
|
|
Jangol
|
77.78
|
74.07
|
55.56
|
59.26
|
66.67
|
81.48
|
66.67
|
66.67
|
70.37
|
74.07
|
|
Jinpung
|
81.48
|
48.15
|
59.26
|
62.96
|
62.96
|
62.96
|
55.56
|
55.56
|
66.67
|
70.37
|
|
Haepum
|
62.96
|
74.07
|
55.56
|
51.85
|
59.26
|
66.67
|
51.85
|
66.67
|
55.56
|
66.67
|
|
Yongpoong
|
70.37
|
74.07
|
70.37
|
66.67
|
66.67
|
74.07
|
74.07
|
66.67
|
70.37
|
81.48
|
|
Muhan
kong
|
Bogwang
|
Samnam
kong
|
Saeal
kong
|
Seonyu
|
Sodam
kong
|
Songhag
kong
|
Alchan
kong
|
Ilmikong
|
Jangmikong
|
|
Pyeongwon
|
70.37
|
59.26
|
55.56
|
59.26
|
59.26
|
66.67
|
77.78
|
66.67
|
48.15
|
55.56
|
|
Cheongja5ho
|
66.67
|
55.56
|
66.67
|
62.96
|
55.56
|
62.96
|
59.26
|
62.96
|
66.67
|
44.44
|
|
Taecheong
|
62.96
|
66.67
|
70.37
|
44.44
|
51.85
|
59.26
|
55.56
|
51.85
|
55.56
|
40.74
|
|
Aram
|
55.56
|
44.44
|
48.15
|
51.85
|
44.44
|
37.04
|
33.33
|
51.85
|
55.56
|
48.15
|
|
Cheongja4ho
|
51.85
|
70.37
|
44.44
|
70.37
|
55.56
|
48.15
|
51.85
|
62.96
|
66.67
|
66.67
|
|
Soyeon
|
59.26
|
55.56
|
51.85
|
62.96
|
55.56
|
55.56
|
44.44
|
62.96
|
59.26
|
51.85
|
|
Daepung2ho
|
55.56
|
66.67
|
40.74
|
81.48
|
66.67
|
66.67
|
55.56
|
74.07
|
55.56
|
70.37
|
|
Daechan
|
55.56
|
66.67
|
48.15
|
66.67
|
66.67
|
59.26
|
62.96
|
66.67
|
62.96
|
70.37
|
|
Miso
|
55.56
|
66.67
|
70.37
|
74.07
|
66.67
|
66.67
|
70.37
|
74.07
|
62.96
|
55.56
|
|
Socheongja
|
55.56
|
66.67
|
62.96
|
59.26
|
74.07
|
66.67
|
55.56
|
59.26
|
62.96
|
62.96
|
|
Duruol
|
55.56
|
66.67
|
62.96
|
74.07
|
81.48
|
66.67
|
62.96
|
59.26
|
70.37
|
70.37
|
|
Haewon
|
51.85
|
48.15
|
51.85
|
62.96
|
55.56
|
48.15
|
44.44
|
62.96
|
51.85
|
51.85
|
|
Taeseon
|
40.74
|
66.67
|
40.74
|
59.26
|
59.26
|
59.26
|
48.15
|
59.26
|
55.56
|
70.37
|
|
Seonpung
|
51.85
|
70.37
|
44.44
|
70.37
|
70.37
|
62.96
|
66.67
|
70.37
|
59.26
|
74.07
|
|
Saegeum
|
51.85
|
62.96
|
51.85
|
70.37
|
62.96
|
62.96
|
51.85
|
62.96
|
59.26
|
66.67
|
|
Cheongmiin
|
48.15
|
59.26
|
48.15
|
74.07
|
59.26
|
66.67
|
62.96
|
59.26
|
62.96
|
62.96
|
|
Jangol
|
62.96
|
81.48
|
55.56
|
66.67
|
74.07
|
66.67
|
77.78
|
66.67
|
62.96
|
70.37
|
|
Jinpung
|
51.85
|
55.56
|
44.44
|
62.96
|
70.37
|
62.96
|
51.85
|
62.96
|
51.85
|
74.07
|
|
Haepum
|
55.56
|
51.85
|
62.96
|
59.26
|
59.26
|
51.85
|
55.56
|
59.26
|
62.96
|
62.96
|
|
Yongpoong
|
62.96
|
66.67
|
77.78
|
74.07
|
74.07
|
81.48
|
70.37
|
66.67
|
62.96
|
62.96
|
|
Jangsu
kong
|
Jangyeop
|
Jangwon
kong
|
Jinmi
|
Jinpumkong
|
Jinpumkong2
|
Cheongdu1
|
Taekwang
kong
|
Hojang
|
Hwangkeum
kong
|
|
Pyeongwon
|
62.96
|
62.96
|
59.26
|
55.56
|
62.96
|
66.67
|
66.67
|
66.67
|
70.37
|
74.07
|
|
Cheongja5ho
|
66.67
|
66.67
|
62.96
|
66.67
|
66.67
|
62.96
|
55.56
|
55.56
|
66.67
|
55.56
|
|
Taecheong
|
62.96
|
62.96
|
66.67
|
55.56
|
70.37
|
59.26
|
51.85
|
51.85
|
62.96
|
59.26
|
|
Aram
|
55.56
|
55.56
|
59.26
|
55.56
|
55.56
|
51.85
|
51.85
|
44.44
|
48.15
|
44.44
|
|
Cheongja4ho
|
66.67
|
74.07
|
70.37
|
59.26
|
59.26
|
62.96
|
55.56
|
77.78
|
59.26
|
62.96
|
|
Soyeon
|
66.67
|
66.67
|
70.37
|
59.26
|
51.85
|
48.15
|
62.96
|
55.56
|
59.26
|
55.56
|
|
Daepung2ho
|
62.96
|
77.78
|
66.67
|
62.96
|
55.56
|
59.26
|
66.67
|
81.48
|
62.96
|
66.67
|
|
Daechan
|
70.37
|
70.37
|
66.67
|
62.96
|
62.96
|
74.07
|
66.67
|
74.07
|
62.96
|
66.67
|
|
Miso
|
62.96
|
77.78
|
66.67
|
77.78
|
70.37
|
74.07
|
74.07
|
59.26
|
77.78
|
74.07
|
|
Socheongja
|
62.96
|
62.96
|
66.67
|
70.37
|
85.19
|
74.07
|
59.26
|
59.26
|
70.37
|
66.67
|
|
Duruol
|
62.96
|
70.37
|
66.67
|
70.37
|
77.78
|
74.07
|
74.07
|
66.67
|
77.78
|
74.07
|
|
Haewon
|
51.85
|
66.67
|
62.96
|
66.67
|
59.26
|
55.56
|
62.96
|
55.56
|
59.26
|
55.56
|
|
Taeseon
|
48.15
|
55.56
|
59.26
|
55.56
|
55.56
|
51.85
|
51.85
|
66.67
|
55.56
|
51.85
|
|
Seonpung
|
66.67
|
74.07
|
70.37
|
66.67
|
59.26
|
70.37
|
70.37
|
77.78
|
66.67
|
70.37
|
|
Saegeum
|
59.26
|
81.48
|
77.78
|
66.67
|
51.85
|
48.15
|
62.96
|
70.37
|
66.67
|
62.96
|
|
Cheongmiin
|
62.96
|
70.37
|
51.85
|
62.96
|
62.96
|
66.67
|
66.67
|
81.48
|
55.56
|
66.67
|
|
Jangol
|
77.78
|
77.78
|
66.67
|
62.96
|
77.78
|
81.48
|
59.26
|
74.07
|
77.78
|
88.89
|
|
Jinpung
|
59.26
|
66.67
|
62.96
|
59.26
|
66.67
|
62.96
|
62.96
|
77.78
|
66.67
|
62.96
|
|
Haepum
|
62.96
|
70.37
|
66.67
|
62.96
|
62.96
|
59.26
|
66.67
|
51.85
|
70.37
|
66.67
|
|
Yongpoong
|
62.96
|
62.96
|
66.67
|
70.37
|
70.37
|
59.26
|
66.67
|
51.85
|
85.19
|
74.07
|
|
Kwangan
kong
|
Namhae
kong
|
Nogchae
|
Dagi
|
Dawonkong
|
Dachae
|
Doremikong
|
Myeongju
namul
kong
|
Bosuk
|
BuKangkong
|
|
Pyeongwon
|
51.85
|
59.26
|
55.56
|
70.37
|
59.26
|
62.96
|
70.37
|
51.85
|
55.56
|
62.96
|
|
Cheongja5ho
|
62.96
|
55.56
|
37.04
|
59.26
|
55.56
|
59.26
|
66.67
|
48.15
|
51.85
|
66.67
|
|
Taecheong
|
51.85
|
51.85
|
48.15
|
55.56
|
66.67
|
55.56
|
48.15
|
66.67
|
48.15
|
55.56
|
|
Aram
|
66.67
|
59.26
|
33.33
|
55.56
|
44.44
|
48.15
|
55.56
|
51.85
|
70.37
|
62.96
|
|
Cheongja4ho
|
70.37
|
62.96
|
44.44
|
59.26
|
70.37
|
59.26
|
59.26
|
62.96
|
51.85
|
66.67
|
|
Soyeon
|
62.96
|
70.37
|
51.85
|
66.67
|
48.15
|
51.85
|
66.67
|
48.15
|
74.07
|
74.07
|
|
Daepung2ho
|
59.26
|
74.07
|
55.56
|
62.96
|
66.67
|
55.56
|
62.96
|
51.85
|
55.56
|
70.37
|
|
Daechan
|
59.26
|
66.67
|
62.96
|
62.96
|
59.26
|
70.37
|
62.96
|
59.26
|
48.15
|
62.96
|
|
Miso
|
51.85
|
66.67
|
55.56
|
62.96
|
74.07
|
55.56
|
62.96
|
51.85
|
62.96
|
70.37
|
|
Socheongja
|
66.67
|
66.67
|
62.96
|
70.37
|
66.67
|
70.37
|
62.96
|
66.67
|
55.56
|
70.37
|
|
Duruol
|
74.07
|
59.26
|
62.96
|
70.37
|
74.07
|
70.37
|
70.37
|
59.26
|
55.56
|
70.37
|
|
Haewon
|
62.96
|
70.37
|
44.44
|
66.67
|
55.56
|
59.26
|
66.67
|
48.15
|
81.48
|
74.07
|
|
Taeseon
|
59.26
|
66.67
|
62.96
|
55.56
|
66.67
|
55.56
|
48.15
|
59.26
|
48.15
|
62.96
|
|
Seonpung
|
55.56
|
70.37
|
66.67
|
66.67
|
62.96
|
74.07
|
66.67
|
62.96
|
51.85
|
66.67
|
|
Saegeum
|
77.78
|
70.37
|
66.67
|
74.07
|
62.96
|
66.67
|
74.07
|
55.56
|
66.67
|
81.48
|
|
Cheongmiin
|
59.26
|
59.26
|
48.15
|
55.56
|
51.85
|
55.56
|
62.96
|
44.44
|
48.15
|
62.96
|
|
Jangol
|
59.26
|
66.67
|
55.56
|
70.37
|
81.48
|
70.37
|
70.37
|
59.26
|
48.15
|
70.37
|
|
Jinpung
|
70.37
|
62.96
|
51.85
|
66.67
|
62.96
|
66.67
|
66.67
|
70.37
|
51.85
|
74.07
|
|
Haepum
|
66.67
|
66.67
|
55.56
|
77.78
|
59.26
|
70.37
|
77.78
|
59.26
|
77.78
|
70.37
|
|
Yongpoong
|
59.26
|
59.26
|
70.37
|
70.37
|
74.07
|
55.56
|
62.96
|
59.26
|
62.96
|
62.96
|
|
Seabyeolkong
|
Seonam
|
Sogang
|
Sorog
|
Somyeongkong
|
Sobaegnamulkong
|
Sowonkong
|
Sojin
|
Sohokong
|
Singang
|
|
Pyeongwon
|
51.85
|
59.26
|
51.85
|
66.67
|
48.15
|
55.56
|
70.37
|
55.56
|
66.67
|
62.96
|
|
Cheongja5ho
|
48.15
|
48.15
|
62.96
|
55.56
|
51.85
|
51.85
|
51.85
|
44.44
|
62.96
|
51.85
|
|
Taecheong
|
51.85
|
59.26
|
51.85
|
59.26
|
48.15
|
55.56
|
55.56
|
40.74
|
51.85
|
48.15
|
|
Aram
|
51.85
|
51.85
|
74.07
|
66.67
|
85.19
|
55.56
|
55.56
|
70.37
|
59.26
|
55.56
|
|
Cheongja4ho
|
55.56
|
55.56
|
55.56
|
62.96
|
59.26
|
59.26
|
66.67
|
59.26
|
70.37
|
59.26
|
|
Soyeon
|
62.96
|
55.56
|
85.19
|
62.96
|
74.07
|
66.67
|
51.85
|
59.26
|
70.37
|
51.85
|
|
Daepung2ho
|
66.67
|
59.26
|
59.26
|
59.26
|
48.15
|
62.96
|
62.96
|
55.56
|
81.48
|
55.56
|
|
Daechan
|
66.67
|
59.26
|
59.26
|
51.85
|
48.15
|
55.56
|
62.96
|
48.15
|
74.07
|
55.56
|
|
Miso
|
59.26
|
59.26
|
51.85
|
66.67
|
48.15
|
70.37
|
55.56
|
48.15
|
74.07
|
48.15
|
|
Socheongja
|
66.67
|
74.07
|
59.26
|
66.67
|
48.15
|
55.56
|
62.96
|
55.56
|
66.67
|
55.56
|
|
Duruol
|
51.85
|
66.67
|
51.85
|
74.07
|
48.15
|
70.37
|
70.37
|
55.56
|
66.67
|
62.96
|
|
Haewon
|
62.96
|
62.96
|
70.37
|
77.78
|
74.07
|
66.67
|
59.26
|
74.07
|
70.37
|
59.26
|
|
Taeseon
|
66.67
|
59.26
|
51.85
|
59.26
|
48.15
|
55.56
|
48.15
|
55.56
|
59.26
|
48.15
|
|
Seonpung
|
70.37
|
62.96
|
55.56
|
55.56
|
44.44
|
59.26
|
66.67
|
51.85
|
77.78
|
59.26
|
|
Saegeum
|
62.96
|
55.56
|
62.96
|
62.96
|
51.85
|
74.07
|
59.26
|
51.85
|
70.37
|
59.26
|
|
Cheongmiin
|
51.85
|
51.85
|
44.44
|
59.26
|
40.74
|
62.96
|
55.56
|
48.15
|
66.67
|
55.56
|
|
Jangol
|
59.26
|
74.07
|
51.85
|
74.07
|
40.74
|
62.96
|
70.37
|
48.15
|
74.07
|
62.96
|
|
Jinpung
|
55.56
|
62.96
|
55.56
|
62.96
|
59.26
|
51.85
|
81.48
|
66.67
|
70.37
|
74.07
|
|
Haepum
|
59.26
|
59.26
|
66.67
|
74.07
|
70.37
|
77.78
|
70.37
|
70.37
|
66.67
|
70.37
|
|
Yongpoong
|
51.85
|
59.26
|
51.85
|
59.26
|
40.74
|
70.37
|
55.56
|
55.56
|
59.26
|
48.15
|
|
Sinhwa
|
Anpyeong
|
Wonkwang
|
Wonhwang
|
Eunhakong
|
Iksannamul
kong
|
Janggi
|
Jonam
|
Paldonamul
kong
|
Pureunkong
|
|
Pyeongwon
|
66.67
|
62.96
|
51.85
|
55.56
|
59.26
|
62.96
|
44.44
|
62.96
|
55.56
|
62.96
|
|
Cheongja5ho
|
55.56
|
51.85
|
55.56
|
51.85
|
55.56
|
66.67
|
40.74
|
51.85
|
59.26
|
59.26
|
|
Taecheong
|
51.85
|
55.56
|
51.85
|
55.56
|
51.85
|
48.15
|
44.44
|
55.56
|
55.56
|
55.56
|
|
Aram
|
51.85
|
55.56
|
81.48
|
62.96
|
51.85
|
55.56
|
59.26
|
55.56
|
48.15
|
48.15
|
|
Cheongja4ho
|
62.96
|
51.85
|
62.96
|
66.67
|
55.56
|
74.07
|
55.56
|
66.67
|
59.26
|
59.26
|
|
Soyeon
|
40.74
|
59.26
|
77.78
|
59.26
|
62.96
|
66.67
|
55.56
|
59.26
|
66.67
|
51.85
|
|
Daepung2ho
|
51.85
|
55.56
|
59.26
|
55.56
|
59.26
|
77.78
|
51.85
|
62.96
|
70.37
|
55.56
|
|
Daechan
|
51.85
|
55.56
|
59.26
|
55.56
|
51.85
|
70.37
|
51.85
|
62.96
|
70.37
|
62.96
|
|
Miso
|
59.26
|
55.56
|
44.44
|
55.56
|
51.85
|
70.37
|
51.85
|
62.96
|
70.37
|
77.78
|
|
Socheongja
|
59.26
|
62.96
|
59.26
|
55.56
|
59.26
|
62.96
|
44.44
|
70.37
|
62.96
|
70.37
|
|
Duruol
|
74.07
|
62.96
|
51.85
|
55.56
|
74.07
|
62.96
|
51.85
|
70.37
|
70.37
|
77.78
|
|
Haewon
|
55.56
|
59.26
|
70.37
|
66.67
|
62.96
|
66.67
|
55.56
|
66.67
|
51.85
|
59.26
|
|
Taeseon
|
44.44
|
48.15
|
59.26
|
55.56
|
51.85
|
55.56
|
59.26
|
62.96
|
48.15
|
48.15
|
|
Seonpung
|
55.56
|
59.26
|
55.56
|
59.26
|
55.56
|
74.07
|
55.56
|
66.67
|
66.67
|
66.67
|
|
Saegeum
|
48.15
|
66.67
|
55.56
|
51.85
|
70.37
|
74.07
|
48.15
|
66.67
|
74.07
|
66.67
|
|
Cheongmiin
|
59.26
|
48.15
|
44.44
|
48.15
|
59.26
|
70.37
|
51.85
|
55.56
|
62.96
|
55.56
|
|
Jangol
|
66.67
|
62.96
|
51.85
|
48.15
|
66.67
|
70.37
|
44.44
|
77.78
|
70.37
|
70.37
|
|
Jinpung
|
70.37
|
66.67
|
70.37
|
59.26
|
62.96
|
74.07
|
55.56
|
66.67
|
59.26
|
59.26
|
|
Haepum
|
66.67
|
70.37
|
66.67
|
62.96
|
74.07
|
62.96
|
59.26
|
77.78
|
62.96
|
70.37
|
|
Yongpoong
|
59.26
|
62.96
|
37.04
|
48.15
|
59.26
|
55.56
|
44.44
|
62.96
|
70.37
|
77.78
|
|
Pungsannamulkong
|
Pungwon
|
Hannamkong
|
Hoseo
|
Geomjeongsaeol
|
Cheongja3
|
W82
|
Enrei
|
PI96983
|
L29
|
|
Pyeongwon
|
55.56
|
77.78
|
66.67
|
51.85
|
62.96
|
70.37
|
62.96
|
70.37
|
55.56
|
55.56
|
|
Cheongja5ho
|
59.26
|
59.26
|
48.15
|
62.96
|
66.67
|
74.07
|
59.26
|
66.67
|
66.67
|
51.85
|
|
Taecheong
|
40.74
|
55.56
|
59.26
|
51.85
|
48.15
|
70.37
|
55.56
|
55.56
|
48.15
|
55.56
|
|
Aram
|
40.74
|
55.56
|
44.44
|
59.26
|
40.74
|
62.96
|
62.96
|
40.74
|
40.74
|
55.56
|
|
Cheongja4ho
|
66.67
|
74.07
|
48.15
|
48.15
|
51.85
|
66.67
|
66.67
|
59.26
|
59.26
|
59.26
|
|
Soyeon
|
51.85
|
51.85
|
62.96
|
62.96
|
44.44
|
66.67
|
74.07
|
51.85
|
51.85
|
66.67
|
|
Daepung2ho
|
62.96
|
62.96
|
59.26
|
44.44
|
55.56
|
62.96
|
70.37
|
62.96
|
70.37
|
62.96
|
|
Daechan
|
62.96
|
62.96
|
66.67
|
44.44
|
55.56
|
55.56
|
70.37
|
55.56
|
70.37
|
62.96
|
|
Miso
|
62.96
|
70.37
|
59.26
|
59.26
|
62.96
|
77.78
|
77.78
|
70.37
|
70.37
|
70.37
|
|
Socheongja
|
55.56
|
62.96
|
66.67
|
51.85
|
55.56
|
55.56
|
62.96
|
55.56
|
70.37
|
55.56
|
|
Duruol
|
62.96
|
77.78
|
66.67
|
66.67
|
70.37
|
62.96
|
70.37
|
70.37
|
62.96
|
62.96
|
|
Haewon
|
51.85
|
66.67
|
55.56
|
62.96
|
51.85
|
66.67
|
74.07
|
44.44
|
51.85
|
66.67
|
|
Taeseon
|
48.15
|
48.15
|
59.26
|
44.44
|
48.15
|
55.56
|
55.56
|
62.96
|
55.56
|
48.15
|
|
Seonpung
|
66.67
|
66.67
|
70.37
|
40.74
|
59.26
|
59.26
|
74.07
|
59.26
|
74.07
|
66.67
|
|
Saegeum
|
74.07
|
59.26
|
77.78
|
62.96
|
51.85
|
66.67
|
81.48
|
51.85
|
59.26
|
74.07
|
|
Cheongmiin
|
62.96
|
70.37
|
59.26
|
51.85
|
62.96
|
62.96
|
62.96
|
70.37
|
70.37
|
55.56
|
|
Jangol
|
70.37
|
85.19
|
66.67
|
51.85
|
55.56
|
62.96
|
77.78
|
62.96
|
70.37
|
70.37
|
|
Jinpung
|
66.67
|
66.67
|
55.56
|
48.15
|
59.26
|
59.26
|
66.67
|
59.26
|
66.67
|
59.26
|
|
Haepum
|
62.96
|
77.78
|
66.67
|
74.07
|
55.56
|
62.96
|
85.19
|
48.15
|
55.56
|
77.78
|
|
Yongpoong
|
62.96
|
62.96
|
66.67
|
66.67
|
70.37
|
70.37
|
62.96
|
77.78
|
70.37
|
55.56
|
|
V94_5152
|
Lee68
|
Kwangkyo
|
Deokyu
|
Sinpaldal
kong
|
Paldalkong
|
Hwaseong
putkong
|
Heugcheong
kong
|
Seoritae
|
Seomoktae
|
|
Pyeongwon
|
74.07
|
44.44
|
66.67
|
59.26
|
44.44
|
59.26
|
66.67
|
55.56
|
66.67
|
59.26
|
|
Cheongja5ho
|
55.56
|
48.15
|
55.56
|
48.15
|
48.15
|
55.56
|
55.56
|
59.26
|
62.96
|
48.15
|
|
Taecheong
|
51.85
|
44.44
|
51.85
|
37.04
|
37.04
|
66.67
|
59.26
|
70.37
|
51.85
|
51.85
|
|
Aram
|
51.85
|
59.26
|
37.04
|
44.44
|
44.44
|
44.44
|
37.04
|
55.56
|
44.44
|
44.44
|
|
Cheongja4ho
|
77.78
|
55.56
|
55.56
|
70.37
|
48.15
|
70.37
|
48.15
|
59.26
|
62.96
|
48.15
|
|
Soyeon
|
55.56
|
55.56
|
55.56
|
55.56
|
62.96
|
48.15
|
40.74
|
66.67
|
48.15
|
62.96
|
|
Daepung2ho
|
66.67
|
51.85
|
66.67
|
74.07
|
59.26
|
59.26
|
44.44
|
62.96
|
59.26
|
66.67
|
|
Daechan
|
66.67
|
51.85
|
66.67
|
74.07
|
66.67
|
66.67
|
51.85
|
62.96
|
51.85
|
59.26
|
|
Miso
|
51.85
|
51.85
|
66.67
|
51.85
|
59.26
|
66.67
|
51.85
|
62.96
|
66.67
|
59.26
|
|
Socheongja
|
59.26
|
51.85
|
66.67
|
59.26
|
51.85
|
59.26
|
51.85
|
77.78
|
44.44
|
66.67
|
|
Duruol
|
66.67
|
59.26
|
81.48
|
59.26
|
51.85
|
59.26
|
66.67
|
70.37
|
59.26
|
66.67
|
|
Haewon
|
62.96
|
55.56
|
48.15
|
55.56
|
55.56
|
48.15
|
40.74
|
59.26
|
55.56
|
55.56
|
|
Taeseon
|
51.85
|
44.44
|
51.85
|
66.67
|
51.85
|
66.67
|
37.04
|
55.56
|
51.85
|
59.26
|
|
Seonpung
|
70.37
|
48.15
|
70.37
|
77.78
|
70.37
|
70.37
|
48.15
|
59.26
|
55.56
|
62.96
|
|
Saegeum
|
70.37
|
55.56
|
70.37
|
62.96
|
70.37
|
55.56
|
48.15
|
66.67
|
62.96
|
62.96
|
|
Cheongmiin
|
59.26
|
51.85
|
59.26
|
66.67
|
59.26
|
51.85
|
51.85
|
48.15
|
66.67
|
59.26
|
|
Jangol
|
66.67
|
51.85
|
74.07
|
59.26
|
51.85
|
66.67
|
59.26
|
70.37
|
66.67
|
66.67
|
|
Jinpung
|
77.78
|
48.15
|
62.96
|
70.37
|
55.56
|
62.96
|
48.15
|
59.26
|
48.15
|
55.56
|
|
Haepum
|
66.67
|
59.26
|
59.26
|
51.85
|
59.26
|
59.26
|
51.85
|
62.96
|
59.26
|
51.85
|
|
Yongpoong
|
51.85
|
51.85
|
81.48
|
44.44
|
44.44
|
51.85
|
66.67
|
77.78
|
66.67
|
66.67
|
|
Orialtae
|
Hanagari
|
Jangdan
baekmok
|
Keumgang
solip
|
Chungbukbaek
|
Kwangdu
|
Baegcheon
|
Saedanbaek
|
Sowon2010
|
Ilpum
geomjeong
kong
|
|
Pyeongwon
|
55.56
|
66.67
|
59.26
|
51.85
|
62.96
|
62.96
|
62.96
|
59.26
|
66.67
|
62.96
|
|
Cheongja5ho
|
66.67
|
62.96
|
48.15
|
55.56
|
59.26
|
51.85
|
74.07
|
62.96
|
55.56
|
51.85
|
|
Taecheong
|
55.56
|
51.85
|
66.67
|
66.67
|
62.96
|
55.56
|
48.15
|
51.85
|
51.85
|
55.56
|
|
Aram
|
55.56
|
29.63
|
44.44
|
44.44
|
40.74
|
33.33
|
40.74
|
44.44
|
51.85
|
48.15
|
|
Cheongja4ho
|
66.67
|
55.56
|
62.96
|
55.56
|
51.85
|
51.85
|
51.85
|
62.96
|
62.96
|
81.48
|
|
Soyeon
|
59.26
|
48.15
|
55.56
|
55.56
|
51.85
|
51.85
|
51.85
|
40.74
|
55.56
|
51.85
|
|
Daepung2ho
|
70.37
|
74.07
|
66.67
|
59.26
|
62.96
|
62.96
|
55.56
|
59.26
|
59.26
|
70.37
|
|
Daechan
|
62.96
|
66.67
|
66.67
|
74.07
|
55.56
|
62.96
|
62.96
|
51.85
|
59.26
|
55.56
|
|
Miso
|
62.96
|
74.07
|
74.07
|
74.07
|
62.96
|
62.96
|
62.96
|
66.67
|
51.85
|
55.56
|
|
Socheongja
|
70.37
|
59.26
|
66.67
|
74.07
|
62.96
|
62.96
|
48.15
|
51.85
|
59.26
|
48.15
|
|
Duruol
|
70.37
|
66.67
|
66.67
|
66.67
|
70.37
|
77.78
|
55.56
|
74.07
|
66.67
|
70.37
|
|
Haewon
|
66.67
|
40.74
|
55.56
|
48.15
|
51.85
|
44.44
|
37.04
|
48.15
|
62.96
|
51.85
|
|
Taeseon
|
55.56
|
66.67
|
59.26
|
51.85
|
55.56
|
48.15
|
48.15
|
51.85
|
51.85
|
62.96
|
|
Seonpung
|
66.67
|
70.37
|
70.37
|
70.37
|
59.26
|
66.67
|
59.26
|
55.56
|
62.96
|
59.26
|
|
Saegeum
|
66.67
|
55.56
|
70.37
|
62.96
|
59.26
|
66.67
|
44.44
|
55.56
|
62.96
|
59.26
|
|
Cheongmiin
|
62.96
|
81.48
|
59.26
|
51.85
|
62.96
|
55.56
|
70.37
|
66.67
|
59.26
|
70.37
|
|
Jangol
|
70.37
|
74.07
|
81.48
|
74.07
|
70.37
|
70.37
|
55.56
|
66.67
|
66.67
|
70.37
|
|
Jinpung
|
74.07
|
62.96
|
55.56
|
55.56
|
66.67
|
59.26
|
44.44
|
62.96
|
70.37
|
59.26
|
|
Haepum
|
62.96
|
44.44
|
59.26
|
59.26
|
55.56
|
55.56
|
40.74
|
59.26
|
74.07
|
55.56
|
|
Yongpoong
|
62.96
|
74.07
|
66.67
|
66.67
|
70.37
|
77.78
|
62.96
|
74.07
|
51.85
|
62.96
|
|
Hanol
|
Socheong2
|
Daeha
|
Daeha_1
|
Uram
|
Hwangkeumol
|
Geomjeong5
|
Cheonsang
|
Heugseong
|
Joyang1
|
|
Pyeongwon
|
74.07
|
55.56
|
66.67
|
62.96
|
66.67
|
59.26
|
51.85
|
74.07
|
55.56
|
55.56
|
|
Cheongja5ho
|
70.37
|
51.85
|
62.96
|
66.67
|
70.37
|
70.37
|
55.56
|
62.96
|
66.67
|
59.26
|
|
Taecheong
|
51.85
|
55.56
|
59.26
|
55.56
|
59.26
|
51.85
|
51.85
|
59.26
|
55.56
|
48.15
|
|
Aram
|
51.85
|
70.37
|
37.04
|
40.74
|
51.85
|
44.44
|
59.26
|
66.67
|
62.96
|
40.74
|
|
Cheongja4ho
|
62.96
|
74.07
|
48.15
|
44.44
|
70.37
|
55.56
|
62.96
|
62.96
|
59.26
|
66.67
|
|
Soyeon
|
55.56
|
59.26
|
48.15
|
51.85
|
55.56
|
55.56
|
77.78
|
70.37
|
74.07
|
51.85
|
|
Daepung2ho
|
66.67
|
70.37
|
51.85
|
55.56
|
74.07
|
66.67
|
66.67
|
66.67
|
62.96
|
62.96
|
|
Daechan
|
66.67
|
55.56
|
59.26
|
62.96
|
66.67
|
59.26
|
59.26
|
59.26
|
62.96
|
62.96
|
|
Miso
|
74.07
|
48.15
|
66.67
|
70.37
|
81.48
|
66.67
|
51.85
|
74.07
|
70.37
|
62.96
|
|
Socheongja
|
66.67
|
55.56
|
59.26
|
62.96
|
59.26
|
66.67
|
44.44
|
59.26
|
55.56
|
55.56
|
|
Duruol
|
81.48
|
62.96
|
74.07
|
70.37
|
66.67
|
74.07
|
51.85
|
66.67
|
62.96
|
62.96
|
|
Haewon
|
62.96
|
59.26
|
40.74
|
44.44
|
62.96
|
55.56
|
62.96
|
77.78
|
59.26
|
51.85
|
|
Taeseon
|
59.26
|
62.96
|
44.44
|
48.15
|
59.26
|
59.26
|
59.26
|
44.44
|
48.15
|
48.15
|
|
Seonpung
|
70.37
|
51.85
|
55.56
|
59.26
|
70.37
|
62.96
|
62.96
|
62.96
|
59.26
|
66.67
|
|
Saegeum
|
62.96
|
51.85
|
55.56
|
51.85
|
62.96
|
62.96
|
77.78
|
70.37
|
59.26
|
74.07
|
|
Cheongmiin
|
66.67
|
55.56
|
51.85
|
55.56
|
66.67
|
66.67
|
59.26
|
66.67
|
70.37
|
62.96
|
|
Jangol
|
66.67
|
55.56
|
59.26
|
55.56
|
66.67
|
66.67
|
44.44
|
66.67
|
62.96
|
70.37
|
|
Jinpung
|
70.37
|
59.26
|
48.15
|
51.85
|
62.96
|
70.37
|
62.96
|
62.96
|
51.85
|
66.67
|
|
Haepum
|
66.67
|
55.56
|
51.85
|
48.15
|
59.26
|
59.26
|
59.26
|
74.07
|
62.96
|
62.96
|
|
Yongpoong
|
74.07
|
55.56
|
81.48
|
77.78
|
74.07
|
74.07
|
44.44
|
66.67
|
62.96
|
62.96
|
|
Cheongyeop1
|
Chamol
|
Jungmo3005
|
Jungmo3006
|
Jungmo3007
|
Neulchan
|
Daol
|
Wonheuk
|
Galmikong
|
Jungmo3003
|
|
Pyeongwon
|
62.96
|
48.15
|
59.26
|
70.37
|
62.96
|
81.48
|
70.37
|
59.26
|
66.67
|
59.26
|
|
Cheongja5ho
|
59.26
|
51.85
|
62.96
|
59.26
|
59.26
|
62.96
|
66.67
|
62.96
|
70.37
|
55.56
|
|
Taecheong
|
55.56
|
48.15
|
59.26
|
62.96
|
62.96
|
59.26
|
55.56
|
51.85
|
74.07
|
59.26
|
|
Aram
|
48.15
|
48.15
|
44.44
|
48.15
|
48.15
|
59.26
|
48.15
|
37.04
|
51.85
|
51.85
|
|
Cheongja4ho
|
66.67
|
66.67
|
62.96
|
74.07
|
66.67
|
62.96
|
59.26
|
55.56
|
62.96
|
70.37
|
|
Soyeon
|
66.67
|
59.26
|
55.56
|
51.85
|
59.26
|
62.96
|
51.85
|
40.74
|
55.56
|
55.56
|
|
Daepung2ho
|
77.78
|
70.37
|
66.67
|
62.96
|
77.78
|
59.26
|
62.96
|
51.85
|
59.26
|
66.67
|
|
Daechan
|
70.37
|
62.96
|
59.26
|
62.96
|
62.96
|
59.26
|
55.56
|
59.26
|
66.67
|
59.26
|
|
Miso
|
62.96
|
62.96
|
74.07
|
70.37
|
70.37
|
74.07
|
70.37
|
59.26
|
74.07
|
74.07
|
|
Socheongja
|
55.56
|
55.56
|
66.67
|
70.37
|
70.37
|
59.26
|
62.96
|
66.67
|
81.48
|
59.26
|
|
Duruol
|
62.96
|
62.96
|
74.07
|
77.78
|
77.78
|
74.07
|
77.78
|
66.67
|
81.48
|
81.48
|
|
Haewon
|
51.85
|
59.26
|
48.15
|
59.26
|
59.26
|
70.37
|
59.26
|
48.15
|
55.56
|
62.96
|
|
Taeseon
|
62.96
|
62.96
|
66.67
|
55.56
|
77.78
|
37.04
|
55.56
|
51.85
|
59.26
|
51.85
|
|
Seonpung
|
74.07
|
66.67
|
62.96
|
66.67
|
66.67
|
62.96
|
59.26
|
62.96
|
62.96
|
62.96
|
|
Saegeum
|
66.67
|
74.07
|
55.56
|
66.67
|
66.67
|
70.37
|
59.26
|
55.56
|
62.96
|
70.37
|
|
Cheongmiin
|
70.37
|
70.37
|
74.07
|
70.37
|
62.96
|
66.67
|
70.37
|
51.85
|
51.85
|
66.67
|
|
Jangol
|
70.37
|
62.96
|
74.07
|
92.59
|
70.37
|
74.07
|
70.37
|
66.67
|
81.48
|
74.07
|
|
Jinpung
|
59.26
|
74.07
|
55.56
|
74.07
|
74.07
|
62.96
|
66.67
|
62.96
|
70.37
|
62.96
|
|
Haepum
|
48.15
|
62.96
|
51.85
|
70.37
|
55.56
|
81.48
|
62.96
|
59.26
|
66.67
|
74.07
|
|
Yongpoong
|
62.96
|
48.15
|
81.48
|
62.96
|
77.78
|
74.07
|
77.78
|
66.67
|
81.48
|
74.07
|
|
Jungmo3004
|
Jungmo3002
|
Socheong
|
Hoban
|
Cheonga
|
Gangil
|
Daewang
|
Haesal
|
Gichan
|
Pyeongwon
|
|
Pyeongwon
|
62.96
|
59.26
|
51.85
|
62.96
|
70.37
|
66.67
|
59.26
|
62.96
|
59.26
|
100.00
|
|
Cheongja5ho
|
59.26
|
55.56
|
62.96
|
51.85
|
66.67
|
55.56
|
44.44
|
59.26
|
37.04
|
66.67
|
|
Taecheong
|
55.56
|
51.85
|
59.26
|
55.56
|
48.15
|
40.74
|
40.74
|
44.44
|
44.44
|
55.56
|
|
Aram
|
55.56
|
44.44
|
51.85
|
40.74
|
55.56
|
44.44
|
40.74
|
55.56
|
25.93
|
48.15
|
|
Cheongja4ho
|
81.48
|
70.37
|
62.96
|
59.26
|
66.67
|
48.15
|
44.44
|
59.26
|
37.04
|
66.67
|
|
Soyeon
|
59.26
|
48.15
|
48.15
|
44.44
|
55.56
|
48.15
|
51.85
|
51.85
|
37.04
|
51.85
|
|
Daepung2ho
|
77.78
|
66.67
|
59.26
|
62.96
|
66.67
|
44.44
|
55.56
|
48.15
|
40.74
|
62.96
|
|
Daechan
|
62.96
|
66.67
|
51.85
|
70.37
|
62.96
|
51.85
|
48.15
|
48.15
|
48.15
|
70.37
|
|
Miso
|
70.37
|
66.67
|
51.85
|
70.37
|
62.96
|
70.37
|
66.67
|
59.26
|
55.56
|
70.37
|
|
Socheongja
|
62.96
|
66.67
|
51.85
|
70.37
|
55.56
|
40.74
|
44.44
|
44.44
|
40.74
|
55.56
|
|
Duruol
|
85.19
|
74.07
|
51.85
|
70.37
|
66.67
|
55.56
|
51.85
|
51.85
|
40.74
|
70.37
|
|
Haewon
|
66.67
|
55.56
|
40.74
|
51.85
|
66.67
|
55.56
|
51.85
|
66.67
|
37.04
|
59.26
|
|
Taeseon
|
62.96
|
51.85
|
66.67
|
55.56
|
59.26
|
33.33
|
51.85
|
44.44
|
40.74
|
48.15
|
|
Seonpung
|
66.67
|
70.37
|
48.15
|
74.07
|
66.67
|
55.56
|
51.85
|
51.85
|
51.85
|
74.07
|
|
Saegeum
|
74.07
|
62.96
|
40.74
|
51.85
|
55.56
|
48.15
|
44.44
|
51.85
|
37.04
|
59.26
|
|
Cheongmiin
|
70.37
|
59.26
|
66.67
|
62.96
|
66.67
|
51.85
|
59.26
|
55.56
|
44.44
|
70.37
|
|
Jangol
|
77.78
|
81.48
|
51.85
|
85.19
|
55.56
|
55.56
|
48.15
|
44.44
|
51.85
|
70.37
|
|
Jinpung
|
66.67
|
70.37
|
55.56
|
66.67
|
66.67
|
40.74
|
44.44
|
59.26
|
37.04
|
66.67
|
|
Haepum
|
70.37
|
66.67
|
37.04
|
55.56
|
62.96
|
66.67
|
55.56
|
62.96
|
48.15
|
70.37
|
|
Yongpoong
|
77.78
|
66.67
|
51.85
|
62.96
|
59.26
|
62.96
|
66.67
|
44.44
|
55.56
|
70.37
|
|
Cheongja
5ho
|
Taecheong
|
Aram
|
Cheongja
4ho
|
Soyeon
|
Daepung
2ho
|
Daechan
|
Miso
|
Socheongja
|
Duruol
|
Haewon
|
|
Pyeongwon
|
66.67
|
55.56
|
48.15
|
66.67
|
51.85
|
62.96
|
70.37
|
70.37
|
55.56
|
70.37
|
59.26
|
|
Cheongja5ho
|
100.00
|
59.26
|
59.26
|
70.37
|
62.96
|
59.26
|
66.67
|
74.07
|
66.67
|
66.67
|
55.56
|
|
Taecheong
|
59.26
|
100.00
|
55.56
|
59.26
|
59.26
|
48.15
|
48.15
|
62.96
|
70.37
|
62.96
|
51.85
|
|
Aram
|
59.26
|
55.56
|
100.00
|
59.26
|
81.48
|
55.56
|
48.15
|
55.56
|
55.56
|
55.56
|
88.89
|
|
Cheongja4ho
|
70.37
|
59.26
|
59.26
|
100.00
|
55.56
|
74.07
|
66.67
|
66.67
|
59.26
|
74.07
|
62.96
|
|
Soyeon
|
62.96
|
59.26
|
81.48
|
55.56
|
100.00
|
66.67
|
59.26
|
59.26
|
59.26
|
59.26
|
77.78
|
|
Daepung2ho
|
59.26
|
48.15
|
55.56
|
74.07
|
66.67
|
100.00
|
77.78
|
70.37
|
62.96
|
70.37
|
66.67
|
|
Daechan
|
66.67
|
48.15
|
48.15
|
66.67
|
59.26
|
77.78
|
100.00
|
70.37
|
77.78
|
62.96
|
51.85
|
|
Miso
|
74.07
|
62.96
|
55.56
|
66.67
|
59.26
|
70.37
|
70.37
|
100.00
|
70.37
|
77.78
|
66.67
|
|
Socheongja
|
66.67
|
70.37
|
55.56
|
59.26
|
59.26
|
62.96
|
77.78
|
70.37
|
100.00
|
77.78
|
59.26
|
|
Duruol
|
66.67
|
62.96
|
55.56
|
74.07
|
59.26
|
70.37
|
62.96
|
77.78
|
77.78
|
100.00
|
66.67
|
|
Haewon
|
55.56
|
51.85
|
88.89
|
62.96
|
77.78
|
66.67
|
51.85
|
66.67
|
59.26
|
66.67
|
100.00
|
|
Taeseon
|
51.85
|
62.96
|
48.15
|
66.67
|
59.26
|
70.37
|
62.96
|
62.96
|
70.37
|
62.96
|
51.85
|
|
Seonpung
|
62.96
|
44.44
|
44.44
|
70.37
|
55.56
|
81.48
|
96.30
|
74.07
|
74.07
|
66.67
|
55.56
|
|
Saegeum
|
62.96
|
59.26
|
59.26
|
70.37
|
77.78
|
74.07
|
59.26
|
66.67
|
59.26
|
74.07
|
70.37
|
|
Cheongmiin
|
74.07
|
40.74
|
40.74
|
74.07
|
51.85
|
77.78
|
70.37
|
70.37
|
55.56
|
70.37
|
51.85
|
|
Jangol
|
59.26
|
70.37
|
48.15
|
74.07
|
51.85
|
70.37
|
70.37
|
77.78
|
77.78
|
85.19
|
59.26
|
|
Jinpung
|
70.37
|
51.85
|
59.26
|
77.78
|
55.56
|
74.07
|
74.07
|
66.67
|
74.07
|
74.07
|
62.96
|
|
Haepum
|
59.26
|
55.56
|
77.78
|
66.67
|
74.07
|
55.56
|
55.56
|
70.37
|
62.96
|
77.78
|
88.89
|
|
Yongpoong
|
66.67
|
62.96
|
40.74
|
59.26
|
51.85
|
62.96
|
55.56
|
77.78
|
70.37
|
85.19
|
51.85
|
|
Taeseon
|
Seonpung
|
Saegeum
|
Cheongmiin
|
Jangol
|
Jinpung
|
Haepum
|
Yongpoong
|
|
Pyeongwon
|
48.15
|
74.07
|
59.26
|
70.37
|
70.37
|
66.67
|
70.37
|
70.37
|
|
Cheongja5ho
|
51.85
|
62.96
|
62.96
|
74.07
|
59.26
|
70.37
|
59.26
|
66.67
|
|
Taecheong
|
62.96
|
44.44
|
59.26
|
40.74
|
70.37
|
51.85
|
55.56
|
62.96
|
|
Aram
|
48.15
|
44.44
|
59.26
|
40.74
|
48.15
|
59.26
|
77.78
|
40.74
|
|
Cheongja4ho
|
66.67
|
70.37
|
70.37
|
74.07
|
74.07
|
77.78
|
66.67
|
59.26
|
|
Soyeon
|
59.26
|
55.56
|
77.78
|
51.85
|
51.85
|
55.56
|
74.07
|
51.85
|
|
Daepung2ho
|
70.37
|
81.48
|
74.07
|
77.78
|
70.37
|
74.07
|
55.56
|
62.96
|
|
Daechan
|
62.96
|
96.30
|
59.26
|
70.37
|
70.37
|
74.07
|
55.56
|
55.56
|
|
Miso
|
62.96
|
74.07
|
66.67
|
70.37
|
77.78
|
66.67
|
70.37
|
77.78
|
|
Socheongja
|
70.37
|
74.07
|
59.26
|
55.56
|
77.78
|
74.07
|
62.96
|
70.37
|
|
Duruol
|
62.96
|
66.67
|
74.07
|
70.37
|
85.19
|
74.07
|
77.78
|
85.19
|
|
Haewon
|
51.85
|
55.56
|
70.37
|
51.85
|
59.26
|
62.96
|
88.89
|
51.85
|
|
Taeseon
|
100.00
|
66.67
|
59.26
|
55.56
|
62.96
|
59.26
|
48.15
|
62.96
|
|
Seonpung
|
66.67
|
100.00
|
62.96
|
74.07
|
74.07
|
77.78
|
59.26
|
59.26
|
|
Saegeum
|
59.26
|
62.96
|
100.00
|
66.67
|
66.67
|
70.37
|
74.07
|
66.67
|
|
Cheongmiin
|
55.56
|
74.07
|
66.67
|
100.00
|
70.37
|
74.07
|
55.56
|
62.96
|
|
Jangol
|
62.96
|
74.07
|
66.67
|
70.37
|
100.00
|
74.07
|
70.37
|
70.37
|
|
Jinpung
|
59.26
|
77.78
|
70.37
|
74.07
|
74.07
|
100.00
|
66.67
|
59.26
|
|
Haepum
|
48.15
|
59.26
|
74.07
|
55.56
|
70.37
|
66.67
|
100.00
|
62.96
|
|
Yongpoong
|
62.96
|
59.26
|
66.67
|
62.96
|
70.37
|
59.26
|
62.96
|
100.00
|










