서언
재료 및 방법
실험재료 및 처리방법
단백질 추출
이차원 전기영동
In-gel Digestion
질량 분석 및 생물정보를 이용한 단백질 동정
결과 및 고찰
과습 처리에 따른 수수 잎의 단백질 발현양상
과습 처리에 따른 수수 잎의 Proteome 동정
적요
서언
수수는 아시아와 아프리카의 여러 나라에서 빵과 죽 같은 음식의 목적으로 55%가 소비되고 있으며, 밀(Triticum aestivum L), 쌀(Oryza sativa L.), 옥수수(Zea mays L.), 보리(Hordeum vulgare L.)에 이어서 세계에서 다섯 번째로 중요한 곡물 중의 하나이다(Yoon et al., 2016). 수수는 고온, 다조를 좋아하고 내건성이 강하여 열대와 그에 준하는 건조지대에서 가장 많이 재배될 뿐만 아니라 흑조위축병에 강한 특성을 가지고 있다. 최근 우리나라 밭작물의 논 재배면적 수요는 증가하고 있다. 동시에 밭작물의 주요 생육 기간인 여름철에 강수량이 집중되는 등, 이상기상에 따라 특정 기간의 강수량이 증가하면서 밭작물의 습해 발생 빈도가 높아지고 있는 상황이다. 현재 여러 연구를 통하여 여러 가지 작물들에 대한 작물재배적지에서의 기준이 정립된 바 있지만, 재배적지 이외의 재배지의 작물의 수량 증수 및 품질 향상 재배에 관한 연구는 미미한 수준이며, 습해의 발생기작 분석 분야의 연구 또한 상대적으로 부족한 실정이다.
프로테오믹스는 기능성 단백질 해석, 단백질의 구조변화 게놈의 기능, 생물기능을 해명하는 연구 분야로, 최근의 프로테옴(proteome)이라는 용어는 특정 기관 혹은 조직에서 특정 스트레스에 발현되는 모든 단백질의 발현에도 적용되고 있다(Woo et al., 2003). 프로테오믹스는 단백질 수준에서 유전자 발현에 대한 특성을 파악하고, 외부환경의 변화에 따른 단백질 발현에 대한 정보를 획득할 수 있는 새로운 기술로서 단백질 발현, 발현 후 수식 및 발현단백질의 기능 해명에 유용한 기술이라 할 수 있다(Pandey and Mann, 2000). 이러한 프로테옴 분석은 이차원전기영동을 통하여 특정 조건에 따라 다르게 발현되는 단백질을 전하와 분자량에 따라 분리한 후 각각의 단백질을 질량분석기로 분석하여 아미노산 서열을 결정하고 이를 바탕으로 단백질 또는 유전체 데이터베이스를 생물정보학 툴로 찾아 단백질의 정체를 확인하는 과정으로 구성된다(Rabilloud, 2002).
2000년대에 들어서 복합단백질의 질량분석이 가능한 다차원 단백질 동정기술(MudPIT)과 구축되어 있는 데이터베이스 및 생물정보 관련 프로그램을 이용하는 생물정보학을 접목하는 방향으로 많은 연구가 수행되고 있다(Kim et al., 2010). 최근 기기의 발달로 2-DE에서 분리된 단백질을 효소로 절단하여 펩티드 질량을 정확히 측정할 수 있으며, peptide mapping에 의한 단백질 동정의 성공률도 높아졌다(Kim, 2014). 대량의 동정된 단백질의 생물정보를 바탕으로 단백질 사이에서의 네트워크 분석을 통해 식물체에 미치는 스트레스의 메커니즘을 분자 수준에서 해석하기 위한 연구가 활발히 진행 중에 있다(Komatsu et al., 2013). 식물에서는 애기장대(The Arabidopsis Genome Initiative, 2000)와 벼의 genome에 대한 정보해석(International Rice Genome Sequencing Project, 2005)이 완성됨에 따라, 이 2가지 모델의 식물을 중심으로 하는 단백질에 대한 연구가 가장 활발하게 진행되고 있다(Kim, 2014).
따라서, 본 연구에서는 과습스트레스에 따른 수수의 논 재배를 과학적으로 판별하기 위하여 수수의 초기생육시기인 3엽기와 5엽기에 논 및 과습 논에서의 단백질 발현양상의 차이를 밝히고자 연구를 수행하였다.
재료 및 방법
실험재료 및 처리방법
본 시험은 2016년 농촌진흥청에서 분양 받은 남풍찰 수수를 이용하여 수수 과습 스트레스 관련 단백질 분석을 위한 재료로 이용하였다. 수수 재배를 위해 종자는 수도용 상토와 충북대학교 부속 농장 밭토양과 1:2 비율로 섞어서 만든 토양을 2.5 ㎏씩 채운 Wagner pot (1/10000단보, 한진)에 pot당 4립씩 직파하였다. 파종이 완료된 pot는 충북대학교 부속 농장 온실에서 재배하였다. 수수 발아 후 5일 뒤, 3엽기에 도달하여 3엽기 과습 처리 실험을 10일간 진행하였다. 또한, 발아 후 10일 뒤에 5엽기에 도달하여 5엽기 과습 처리 실험을 10일간 진행하였다. 각각의 처리구에 따른 대조구를 나누어 처리하였다. 처리구의 경우 수수가 심어져 있는 Wagner pot를 더 큰 통에 넣은 후에 pot 밑면이 급수구로부터 수분이 공급되어 토양 표면 10 ㎝ 깊이까지 포화 되도록 처리하였다. 처리기간 10일 동안 매일 토양수분량 측정을 실시하여 대조구에서는 토양수분함량을 평균 25%로 유지하였고, 처리구에서는 평균 58%로 유지하였다. 실험 재료는 과습 처리 10일 후에 각각의 대조구와 처리구에서 수수 잎을 채취하였으며, 채취 후 즉시 액체 질소에 담가 두었던 50 ㎖ 튜브에 넣은 후 곧바로 액체 질소에 동결시켜 시료의 변성을 최소화시켰다.
단백질 추출
단백질 추출에서 효과적인 액체질소를 이용한 세포벽 파쇄 그리고 불순물 제거를 위한 TCA 침전법을 이용하여 수수 잎에서의 단백질 추출을 시행하였다. 50 ㎖ 튜브에 10% TCA와 0.07% 2-Mercaptoethanol을 섞어 만든 용액을 10 ㎖ 채운 후 곱게 갈린 sample 시료를 넣은 후 수 초간 voltexing해 주었다. 1시간 동안 15분마다 voltexing한 후 원심분리(12,000g, 4℃, 20분)하여 상등액을 제거하였다.
상등액 제거 후 acetone으로 희석한 0.07% 2-Mercaptoethanol 용액을 1.5 ㎖ 첨가 후 다시 voltexing, 원심분리(20,000 g, 4℃, 5분)를 실시하였으며 세척 후 speed vacuum centrifuge를 사용하여 잔여 에탄올이 모두 증발되어 제거될 때까지 10분 정도 건조 시켰다. 건조된 시료에 lysis buffer [8M Urea, 2% NP-40, 2% Ampholine (pH 3-10), 5% 2-Mercaptoethanol, 5% Polyvinylpyrrolidone (PVP-40)]를 추가하여 1시간 동안 상온에서 voltexing하여 반응시켜 준 후 원심분리(20,000 g, 25℃, 20분)하여 상층액을 취한 후 Brad Ford 분석을 이용하여 단백질 농도를 결정하였다.
이차원 전기영동
일차원 전기영동(one-dimentional electrophoresis : 1-DE)은 pI (isoelectric point)를 이용한 전기영동으로, 11 ㎝ straw gel [Urea, 30% acrylamide, 10% NP-40, Ampholine (pH 3-10), 10% APS, TEMED] 위에 시료를 주입하여 전기영동 하였다. 전압은 150V에서 1시간, 300V에서 1시간, 500V에서 16시간으로 전압을 증가 조정한 후 전기영동 하였다.
2차 전기영동 분석을 위해서 12% separating gel [1.5M Tris (pH6-8), 30% acrylamide, 10% APS, 10% SDS, TEMED]과 5% Staking gel [0.5M Tris (pH6-8), 30% acrylamide, 10% APS, 10% SDS, TEMED]에 loading 시킨 후 Coomassie brilliant blue (CBB G-250)로 염색해주었다. 모든 실험은 3반복으로 실행되었으며, 염색 후 각각의 gel은 스캐너(HP Scanjet G4010, USA)를 이용하여 스캔했다. 또한, 각각의 2D-gel image analysis는 Progenesis samespot program (Nonlinear Dynamics, Newcastle-upon-Tyne, UK)을 사용하여 분석하였다.
In-gel Digestion
이미지 분석 결과를 바탕으로 하여 각각의 gel에서 단백질 스팟들을 선정하였다. In-gel digestion은 In-Gel Tryptic Digestion Kit (Thermo SCIENTIFIC)을 사용하여 시행하였다. 각각의 gel에서 선정된 단백질 스팟들을 2-D gel에서 메스를 이용하여 추출해 내어 band를 1×1 ㎜ 크기로 절단한 후 600 ㎕ tube에 옮겨주었다. 겔이 옮겨진 튜브에 샘플의 탈색을 위해 destaining buffer [50% ACN/20 mM ammonium bicarbonate]를 200 ㎕ 채운 후 37℃에서 30분간 shaking하였으며 염색된 부분이 겔에서 완전히 사라질 때까지 2∼3번 실시하였다. 색이 완전히 빠진 튜브에 reduction buffer {Digestion buffer [ultrapure water/25mM ammonium bicarbonate]/50 mM TCEP (Tris[2-carboxyethyl]phosphine)}을 30 ㎕ 첨가한 후, 60℃에서 10분간 반응시켜준 다음 alkylation buffer [10% iodoacetoamide/5X digestion buffer] 30 ㎕를 tube에 첨가한 후 상온에서 1시간 반응시켜주었다. Alkylation buffer를 튜브에서 완전히 제거해준 후 washing을 위해 destaining buffer 200 ㎕를 튜브에 첨가 후 37℃에서 15분간 shaking incubation 하였다.
각각의 튜브에 100% ACN 50 ㎕를 첨가한 후 15분간 반응시킨 후 15분 정도 건조시켰다. 튜브에 activated trypsin {Working trypsin[trypsin stock (1 ㎍/㎕-Trypsin/Trypsin Storage Solution)/10X ultrapure water]/10X digestion buffer} 10 ㎕를 첨가한 후 상온에서 15분간 배양하여 겔 조각을 swell 시켜주었다. 그 후 digestion buffer 25 ㎕를 첨가한 후, 30℃에서 overnight시켜 shaking incubation하였다. 새로 준비한 600 ㎕ 튜브에 겔 조각을 제외한 상등액 만을 조심스럽게 옮겨준 다음 speed vacuum centrifuge을 이용하여 완전히 건조 시켜 최종 질량 분석을 위한 시료로 사용하였다.
질량 분석 및 생물정보를 이용한 단백질 동정
단백질 질량 분석은 surveyor HPLC system과 nano-ESI 소스가 장착된 Finningan LTQ-FT-ICR 질량 분석기(Therm Electon, Bremen, Germany)로 구성된 Nano-LC/MS 시스템으로 수행되었다. 샘플들의 염을 모두 제거한 후 각각의 샘플 10 ㎕를 auto sampler (Surveyor)를 사용하여 C18 trap column (I.D., 300 ㎕, length 5 ㎜, particle size 5 ㎕, LC Packings)에 주입한 후 20 ㎕/min의 flow rate로 농축하였다. 이후 MS와 MS/MS acquisition을 반복하며 data-dependent mode로 작동하였고, 실험 조건은 2.2kV spray voltage, 220℃ ion transfer tube temperature, 1.3 millitors collision gas pressure로 모두 세팅하였다. MS/MS auquisition에서 ion 검출은 500 counts를 한계점으로, 활성화는 q=0.25, 활성화시간은 30 ms로 설정하였다. LTQ- FT-ICR MS를 이용하여 알아낸 단백질 펩티드의 MS spectra data값은 먼저 MASCOT ver 2.0 (Matrix Science, UK)의 데이터베이스에서 검색하였으며, 펩티드 오차범위는 50 ppm, fragment ion 오차 범위는 0.5 DA로 설정하였다. 그 결과를 토대로 수집한 protein description과 peptide sequence 정보를 PIR (http://pir.georgetown.edu)을 이용하여 protein sequence를 검색하였다. 최종 확인된 수수 잎의 단백질은 분자량과 pI2가지 항목을 통해 각각의 특성에 따라 분류하여 정리하였다.
결과 및 고찰
과습 처리에 따른 수수 잎의 단백질 발현양상
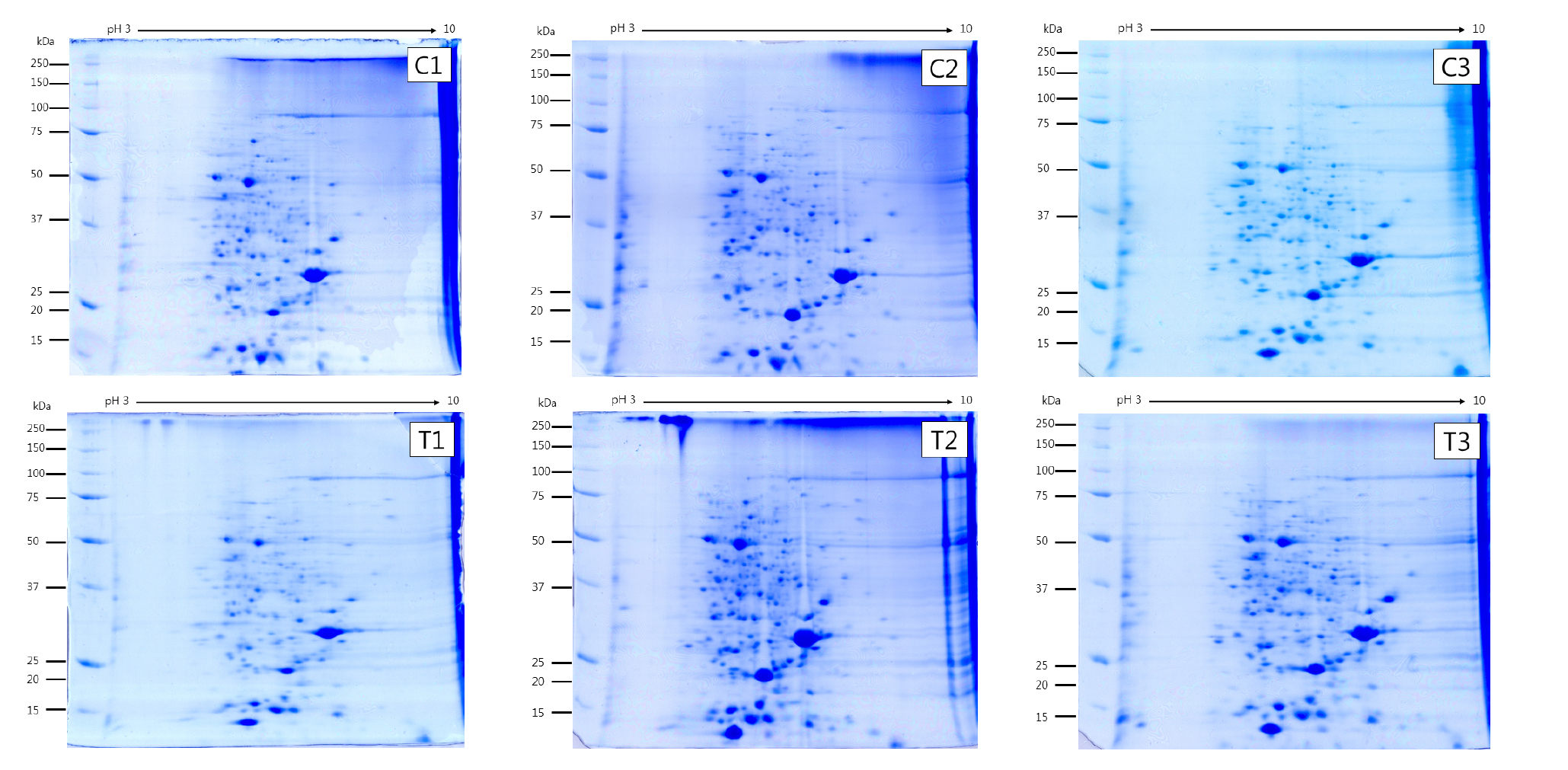
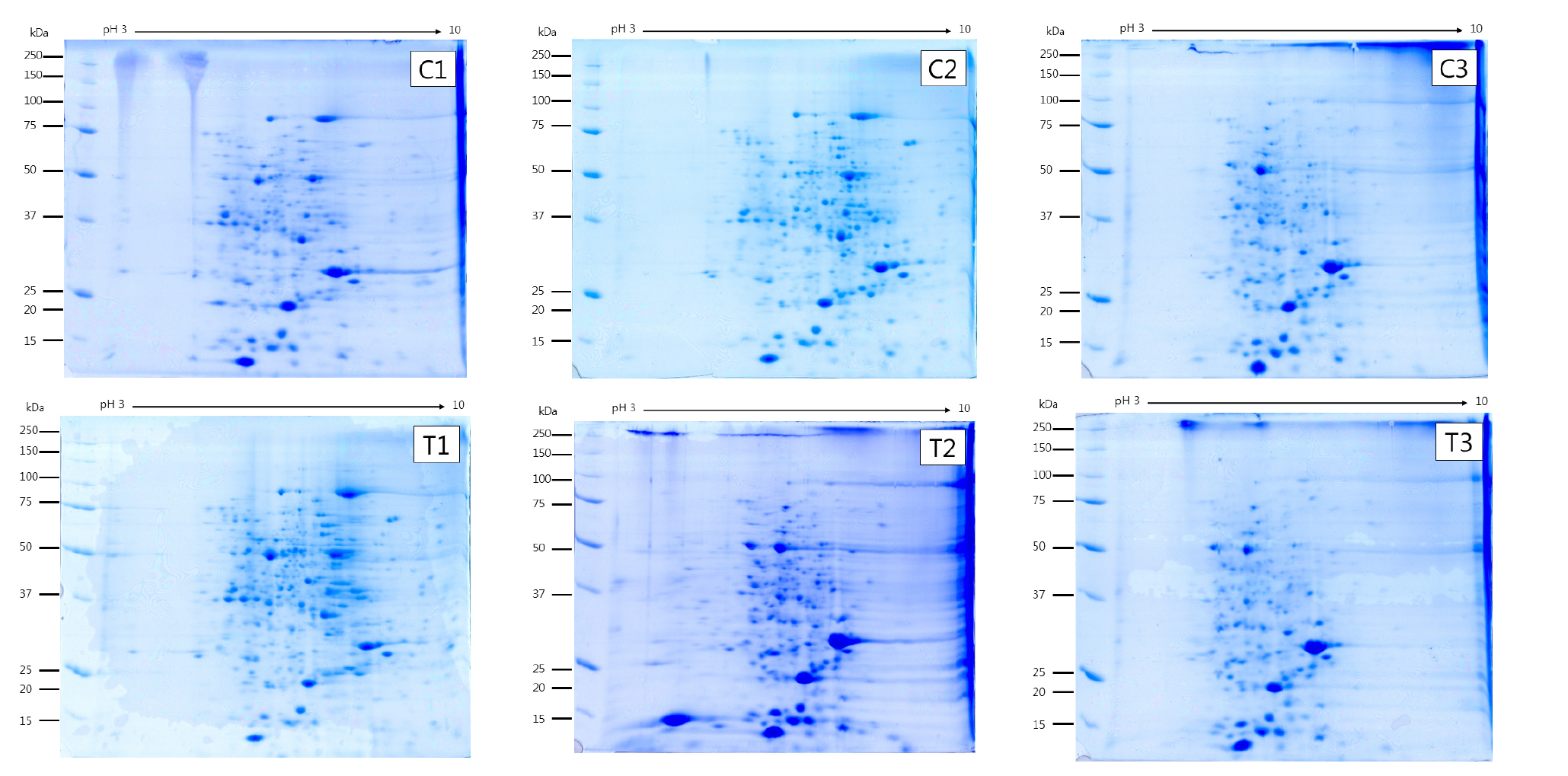
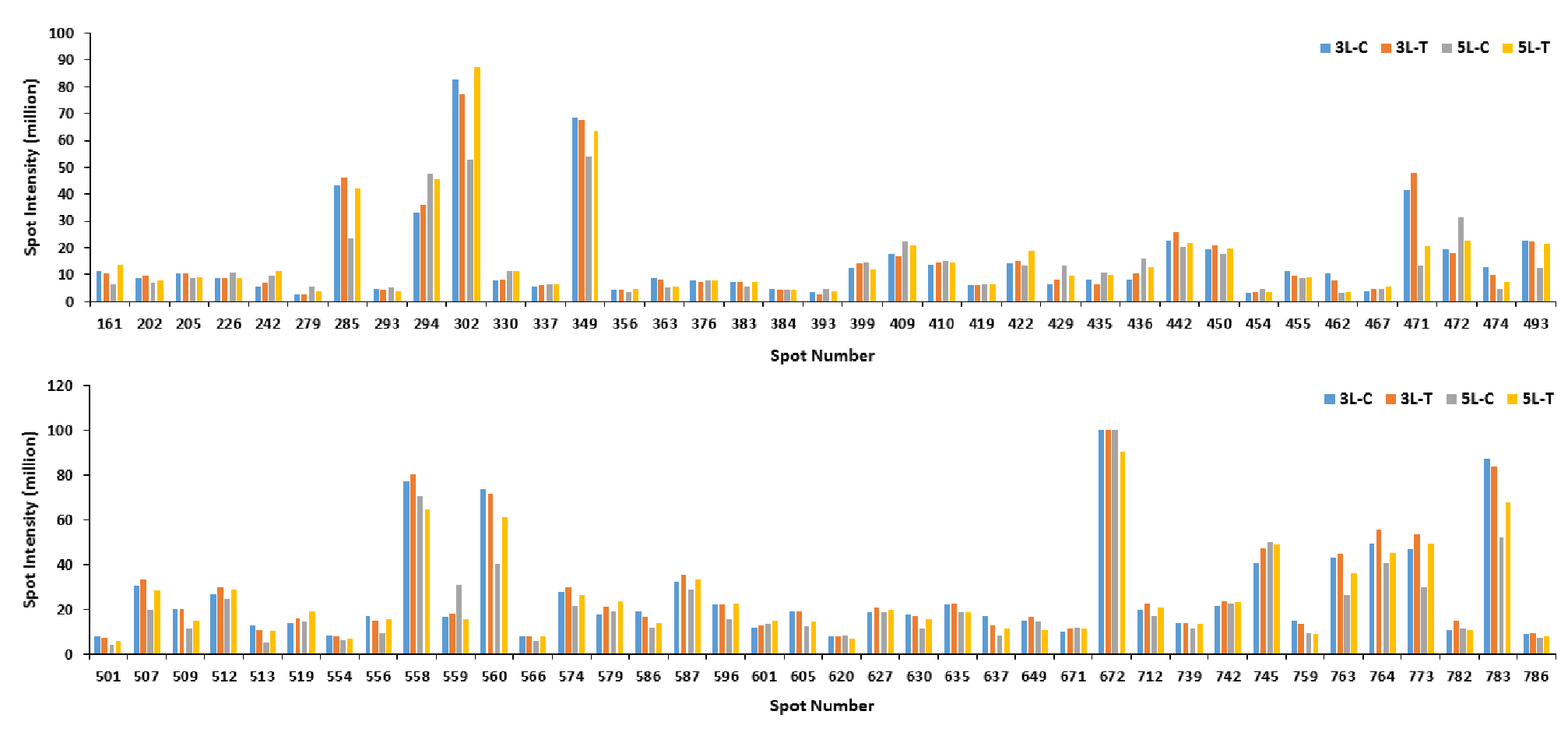
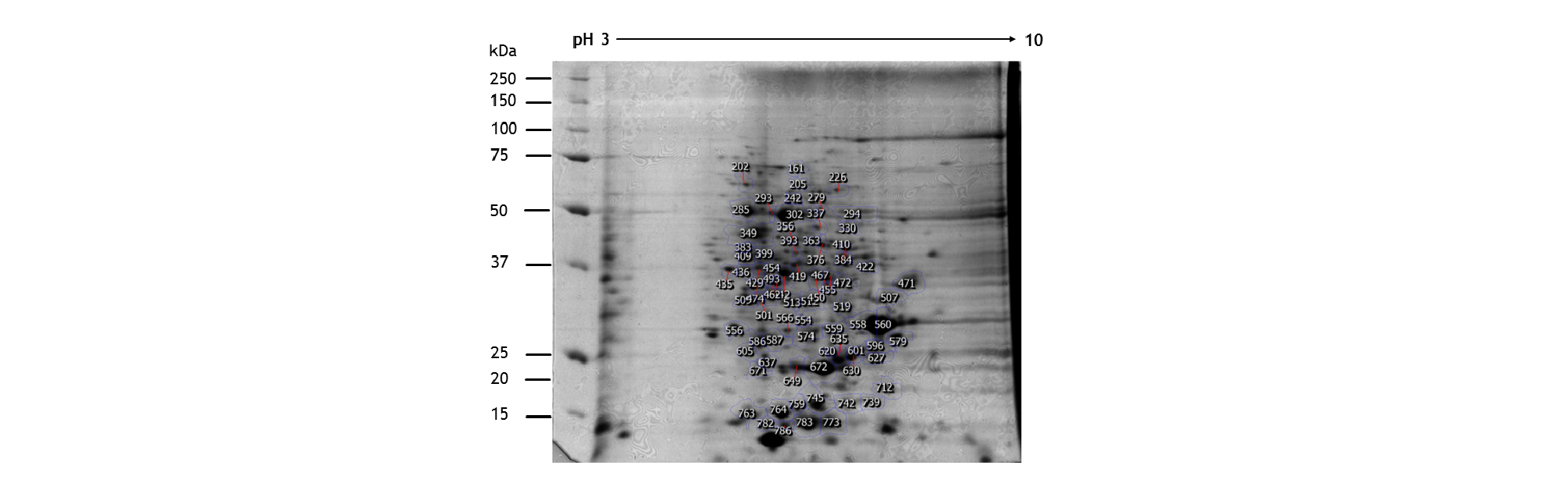
수수의 3엽기에 토양의 과습 처리 10일 후 잎에서부터 추출한 단백질 발현 양상은 Fig. 1과 같고, 5엽기에서의 단백질 발현 양상은 Fig. 2와 같다. 발현된 수많은 단백질 스팟을 Progenesis samespot software program을 이용하여 이미지 분석을 통해 375개의 단백질 스팟으로 구분했다. 구분된 375개의 단백질 스팟 중 서로 다른 양상을 보이며 비교 가능한 단백질 스팟 74개를 선택하여 동정한 결과 실험구 중 토양 과습 처리구에서 20개의 단백질 스팟들에서 up-regulated 된 반면에, 5개의 단백질 스팟들에서는 down-regulated 된 것을 확인할 수 있었다(Fig. 3). Khatoon et al. (2012)은 콩 묘목을 과습처리 후 동정한 결과 과습처리에서 무처리에 비해 4개의 단백질 스팟들에서 down-regulated 된 것을 확인하였다. 이미지 분석 시 기준이 된 reference gel을 질량 분석기로 분석, 동정하였고, 동정결과 각각의 스팟은 구획을 나누어 numbering하여 표시하였다(Fig. 4).
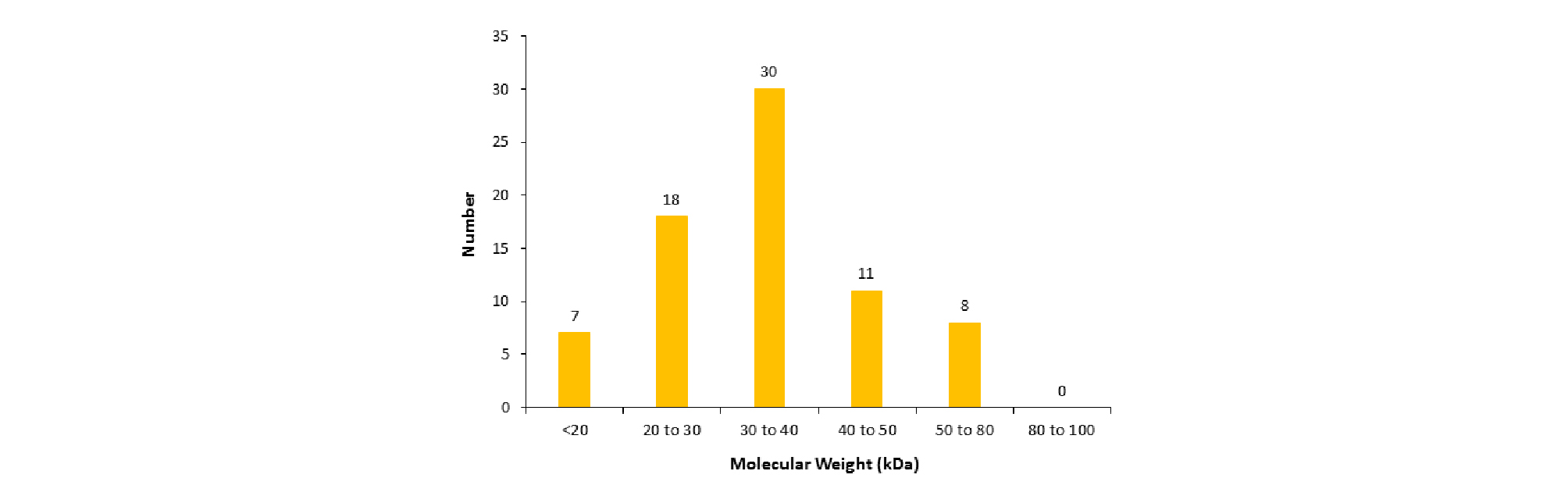
분자 크기 별로 단백질들을 비교한 결과 모두 분자량이 20 kDa∼80 kDa에 속하는 것을 확인하였고, 그 중에 무게가 20 kDa∼40 kDa 크기에 속한 단백질들이 64%를 차지하였으며, 중 분자 범위(50 kDa∼80 kDa)의 경우에는 약 10% 정도 분포하는 것으로 나타났다(Fig. 5).
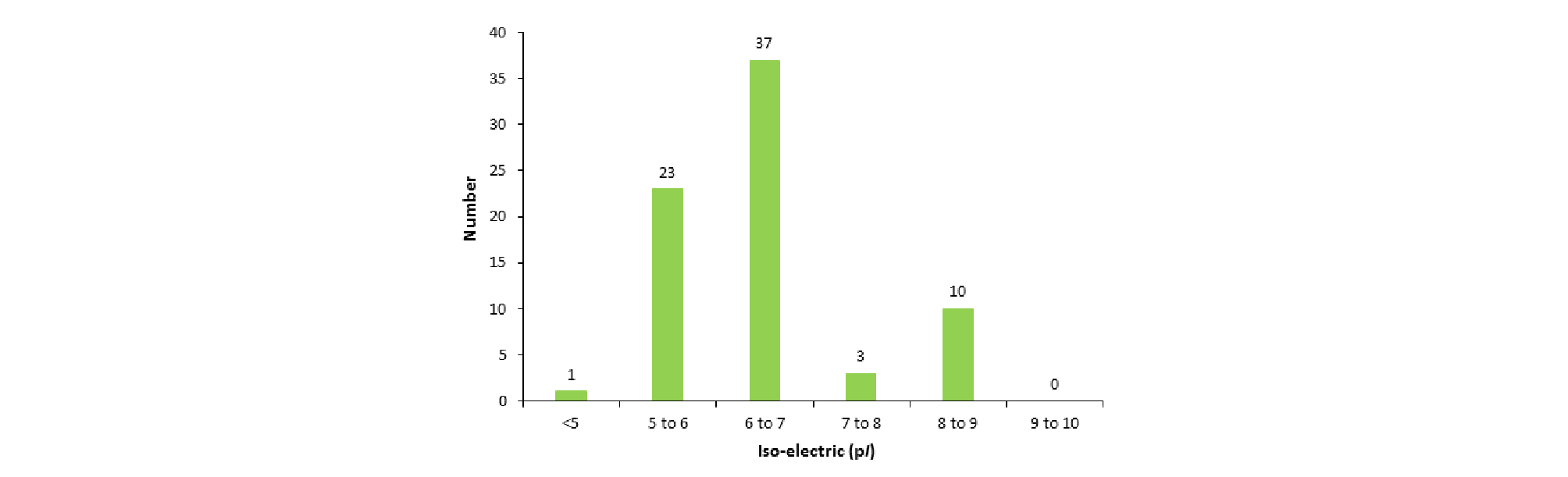
등전점(pI) 또한 배수성에 관계없이 pI 6∼7 사이에서 약 50%로 가장 많은 분포를 보였으며, pI 5∼6 에서도 약 31%로 두 번째로 많은 단백질 분포를 보여 pI5∼7 사이의 단백질들이 약 81% 이상을 차지하였다. 이것으로 보아 약산성에서 중성을 띈 단백질들이 염기성을 띈 단백질 보다 수수 잎에 많이 존재함을 알 수 있었다(Fig. 6). 애기 장대는 대부분 10 kDa∼50 kDa의 작은 분자량을 가진 단백질이 가장 많았으며, 염기성 단백질이 많이 포함되어 있다고 보고하여(Millar et al., 2005) 수수 잎의 분자량에 따른 분포와는 유사한 결과를 보였으나 등전점에 따른 분포는 염기성 보다 산성 단백질이 많이 포함되어 차이가 있음을 나타냈다.
과습 처리에 따른 수수 잎의 Proteome 동정
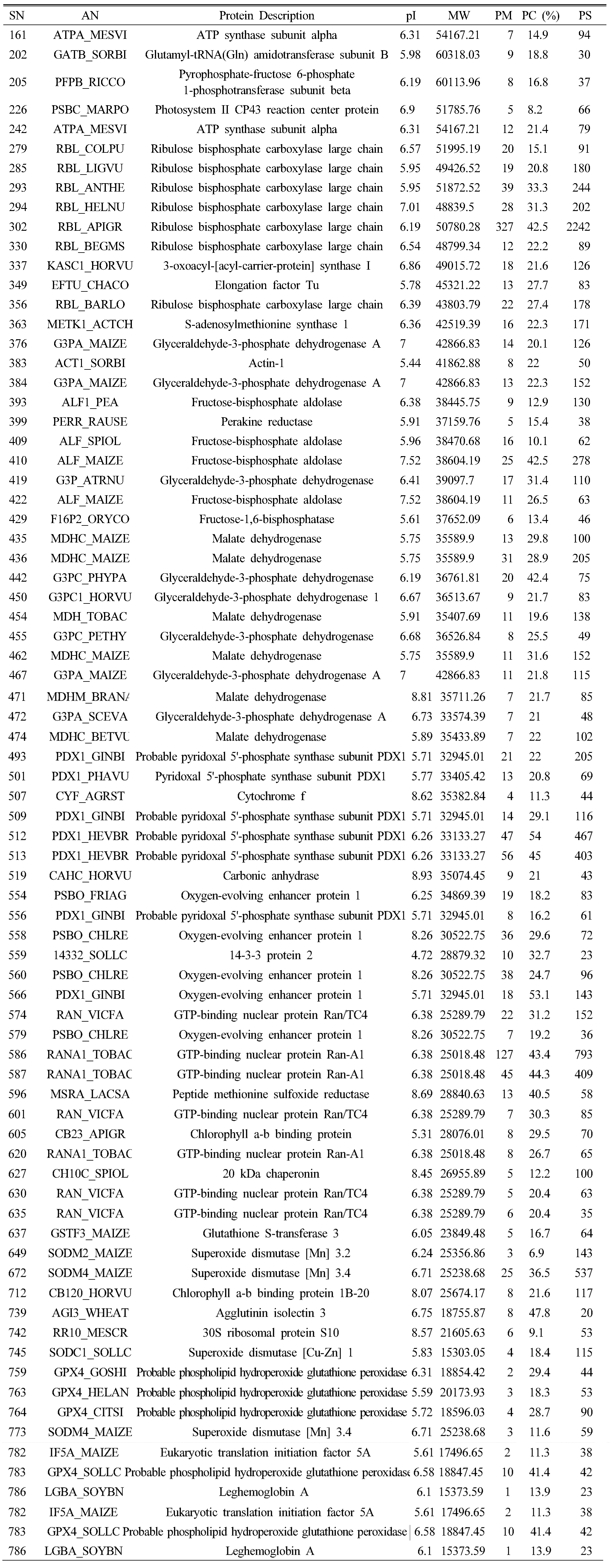
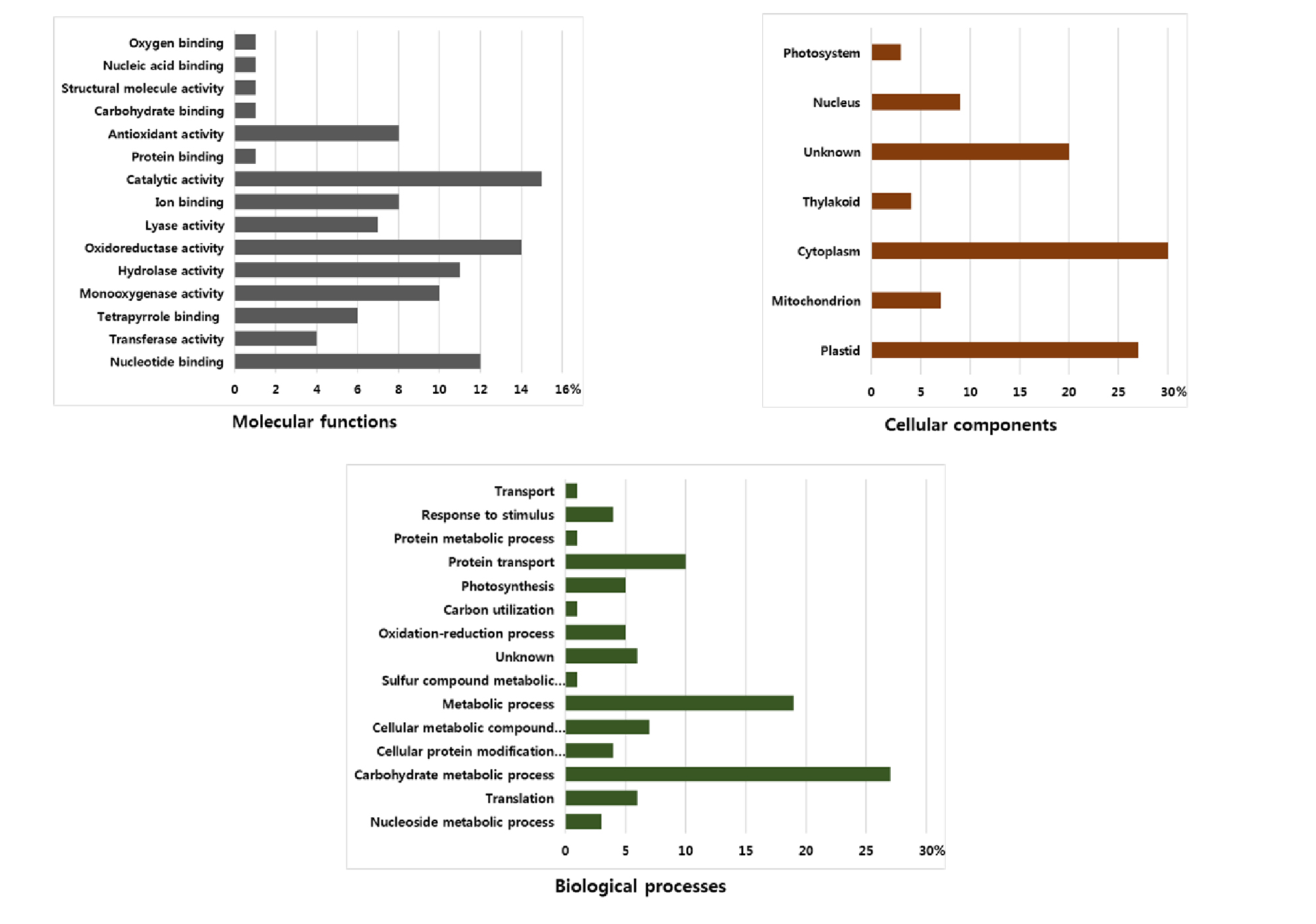
LTQ-FT-ICR MS를 이용하여 토양의 과습 처리 10일 후 수수 잎에서 추출된 단백질들을 동정하였다. 총 74개의 단백질을 동정하였으며, 이렇게 동정된 단백질들을 식별하여 분류한 것은 Table 1과 같다. 그리고 확인된 74개의 단백질들을 molecular function, cellular component, biological process를 기준으로 분류한 것이 Fig. 7이다. Molecular function에서는 촉매활성(catalytic activity)와 산화환원효소활성(oxidoreductase activity)가 각각 15%, 14%로 가장 많이 확인 되었으며 cellular component에서는 세포질, 색소체가 각각 30%, 27%로 가장 많이 확인되었고, biological process에서는 탄수화물 대사과정(carbohydrate metabolic process), 대사과정(metabolic process) 가 각각 27%, 19%로 가장 많은 양을 차지하였다. 탄수화물 대사과정에 작용되는 단백질들이 가장 많이 발견되는 것으로 보아 수수의 3엽기와 5엽기에 과습 스트레스가 탄수화물 대사과정에 많은 영향을 주는 것으로 판단되었으며, 이 외에도 아직 그 기능이 밝혀지지 않은 단백질이 5%가 나타나는 등 여전히 기능 해석이 필요한 단백질이 존재하는 것으로 생각되었다.
Table 1. List of identified differentially expressed proteins from leaves of Sorghum bicolor L. using LTQ-FTICR MS |
|
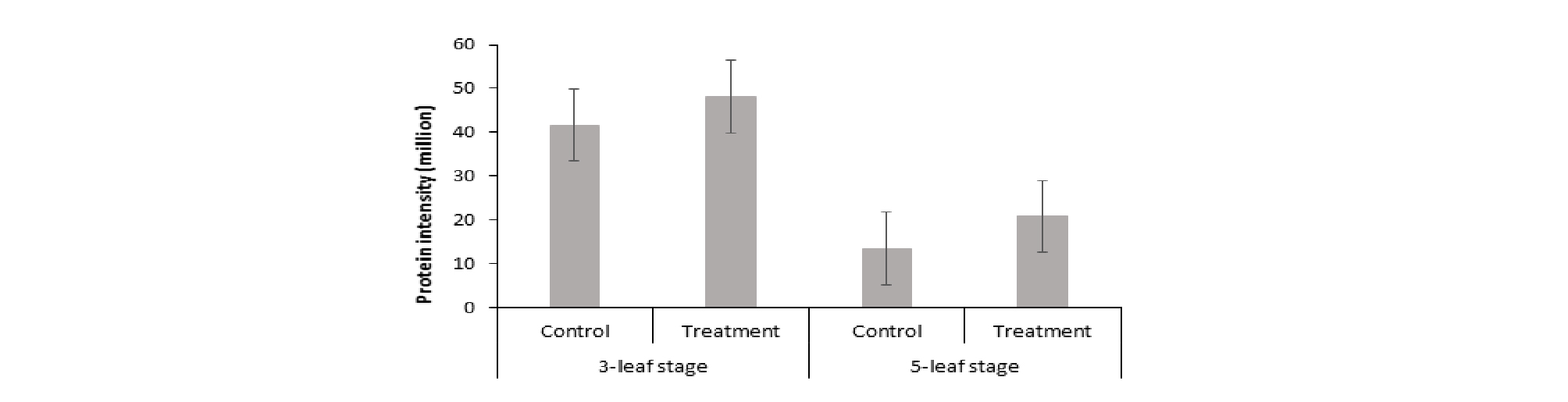
스팟 번호 471인 Malate dehydrogenase는 MDH gene을 가지는 단백질로 Malate dehydrogenase의 활성 변화 정도(Crawford, 1978)는 과습 상태에 따라 극적으로 변화하기 때문에 malate dehydrogenase는 과습 스트레스에 대한 식물 반응의 지표로 사용되어왔다(Daugherty and Musgrave, 1994). 효소학적으로 이 단백질은 Glucose가 EMP, TCA를 거쳐 산화되는 전 과정 중에 하나인 L-말산(Malate)을 NAD+에 의해 가역적으로 탈수소하여 옥살로아세트산(Oxaloacetate)과 NADH를 생성하는 반응을 촉매하는 효소이다((S)-malate + NAD+ = oxaloacetate + NADH). 즉, Malate dehydrogenase가 glucose의 전체 산화과정에서 마지막 NADH2를 생성하는데 촉매 역할을 하는 것이다(Rocha et al., 2010). 생물계에 널리 분포하고, 고등동물에서는 미토콘드리아와 세포질에 같은 기능을 하는 다른 종류의 효소가 존재하는데 주로 미토콘드리아에 존재하면서 molecular function으로는 미토콘드리아탈수소 효소 활성(L-malate dehydrogenase activity)를 하는 역할을 가지고 있으며, 세포기작으로는 탄수화물 대사과정, 말산염 대사과정(malate metabolic process), 트리카르복시산회로(tricarboxylic acid cycle) 과정에서 이용되는 단백질이다.
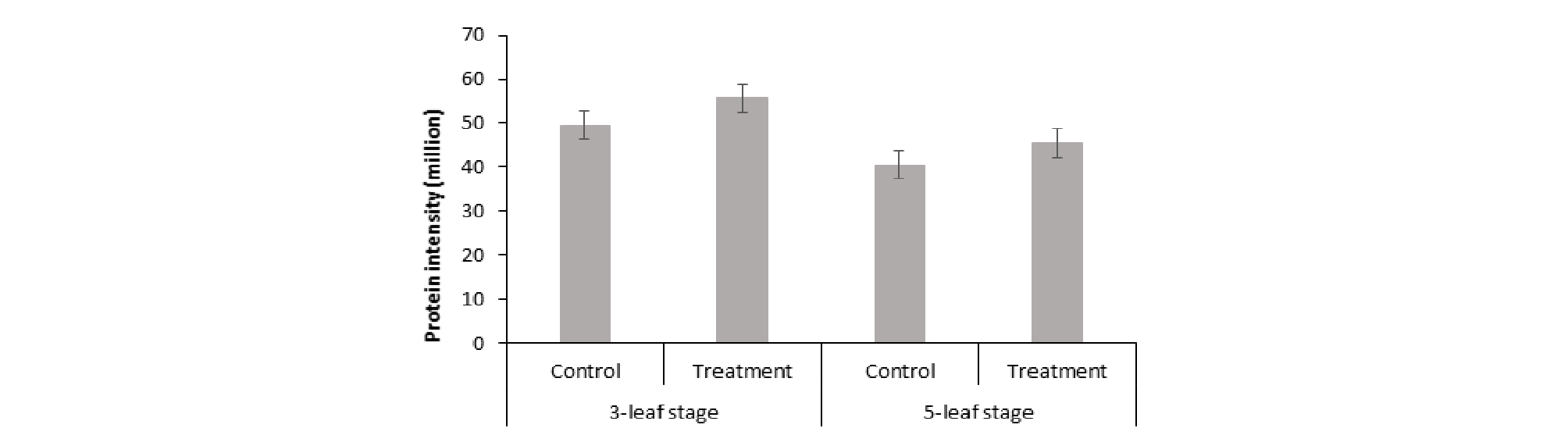
이 단백질을 대조구와 처리구 별로 비교한 그림은 Fig. 8과 같다. 3엽기와 5엽기 모두에서 대조구보다 실험구에서 더 많이 발현되는 양상을 보였는데, 이는 과습 스트레스를 받았을 때, antistress 기능을 하는 단백질로 발현함으로써 그 피해를 줄이려는 경향을 보였다고 사료된다. 또한, 5엽기에서보다 3엽기에서의 발현 양이 대폭 증가되어 있는 것을 보아 어린 시기에 작물에서 말산탈수소효소(malate dehydrogenase)가 더 많이 발현되는 양상을 보임을 확인할 수 있었다.
스팟 번호 579인 Oxygen-evolving enhancer protein 1은(산소전개증폭단백질) PSBO 유전자를 가지는 단백질로서 물 분해의 주요 사이트인 망간 클러스터를 안정화 하는 역할을 하는 단백질로 알려져 있다. 이 단백질들은 물의 분해에서 기능하는 네 개의 Mn2+ 이온들을 결합시키고 높은 산소 발생률을 위한 환경을 유지하는 기능을 가지고 있다(Hoganson and Babcock, 1997). 엽록체 틸라코이드막(Chloroplast thylakoid membrane), 막의 외인성 성분(extrinsic component of membrane), 막의 필수구성요소(integral component of membrane), 광화학계 Ⅱ 산소 발생 복합체(photosystem Ⅱ oxygen evolving complex)에 주로 존재한다. Molecular function으로는 칼슘이온 결합 역할을 하며, biological process에서는 광합성, 광화학계 Ⅱ 안정화(photosystem Ⅱ stabilization) 과정에서 이용된다. 광합성에 주된 작용을 하는 단백질로서 과습 스트레스에 의하여 영향을 받는 것으로 생각되었다. 이 단백질의 발현양의 비교는 Fig. 9와 같다. 대조구에서보다 실험구에서 많이 발현되는 것으로 보아, 과습 스트레스에 의해서 광합성에 부정적인 영향을 받아 부족한 광합성 양을 채우기 위해 실험구에서 이 단백질이 더 많이 발현된 것으로 사료된다.
또한, oxygen-evolving enhancer protein 1은 oxygen- evolving complex 중의 하나이기도하다. 물분해복합체(the water-splitting complex)라고도 알려져 있는 산소전개복합체(the oxygen-evolving complex)는 광합성의 명반응 동안 물의 광산화와 관련된 수산화 효소이다. Oxygen-evolving complex의 도움으로 photosystem II의 반응 중심의 엽록소에서 물 분자를 산화시켜 전자를 제거하고, 결과적으로 총 2개의 물 분자가 4개의 양성자와 4개의 전자 그리고 하나의 산소 분자로 분열된다(Yachandra et al., 1993). 이것은 membrane-lumen interface에서 photosystem II의 핵심 단백질의 4가지 핵심 단백질로 둘러싸여 있다. 1970년부터 Kok에 의하여 널리 인정받은 이론을 바탕으로, 이 복합체는 S0∼S4까지 5가지 상태로 존재할 수 있으며, 광화학계(photosystem) Ⅱ에 의해 포획된 광자(photons)는 system을 S0에서 S4로 이동시킨다. 이 중 S4는 불안정하며 물속에서 발생하는 산소와 반응한다. 현재, 이 복합체의 메커니즘은 완전히 밝혀진 상태가 아니기 때문에 더 많은 연구가 필요한 실정이다.
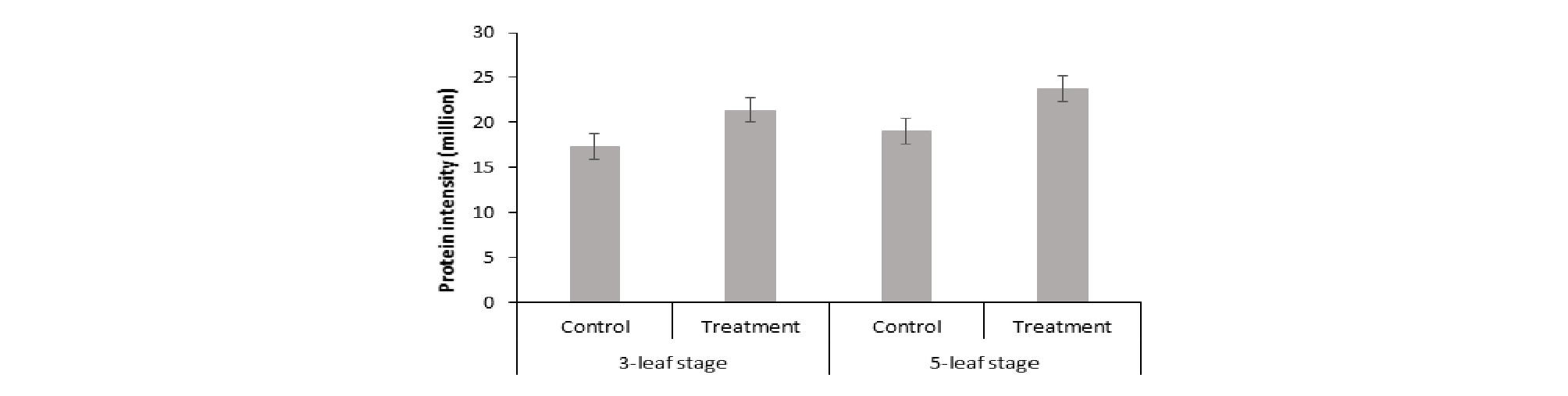
스팟 번호 442인 Glyceraldehyde-3-phosphate dehydrogenase은 GAPC 유전자를 가지고 있는 단백질로서, 해당과정경로의 첫 번째 단계에서 D-glyceraldehyde 3-phosphate를 3-phospho- D-glyceroyl phosphate로 변환시킬 때(D-glyceraldehyde 3-phosphate + phosphate + NAD+ = 3-phospho-D-glyceroyl phosphate + NADH) 촉매로 작용하는 주요 효소이다(Ahsan et al., 2007). 이러한 반응에는 보조 효소Ⅰ(DPN) 또는 Ⅱ (TPN) 및 무기 인산을 필요로 하는데, 식물의 효소인 경우에는 보조 효소 Ⅱ를 필요로 한다. 세포 내 ATP level과 탄수화물 대사의 유지 관리를 위한 필수적인 key enzyme이다. 세포질에 존재하면서 molecular function으로는 glyceraldehyde-3-phosphate dehydrogenase (NAD+) (phosphorylating) activity, NAD binding, NADP binding이 있으며, biological process로는 포도당 대사과정, 당분해 과정에서 이용된다.
Glyceraldehyde-3-phosphate dehydrogenase의 경우 대조구와 과습 처리구의 단백질 발현양 비교는 Fig. 10과 같다. 그림에서 보듯이 대조구에 비해서 과습 처리구에서 발현양이 다소 증가한 것을 볼 수 있다. 이를 토대로 과습 스트레스가 세포 내 탄수화물 대사과정에 영향을 미친다는 것을 확인할 수 있었다.
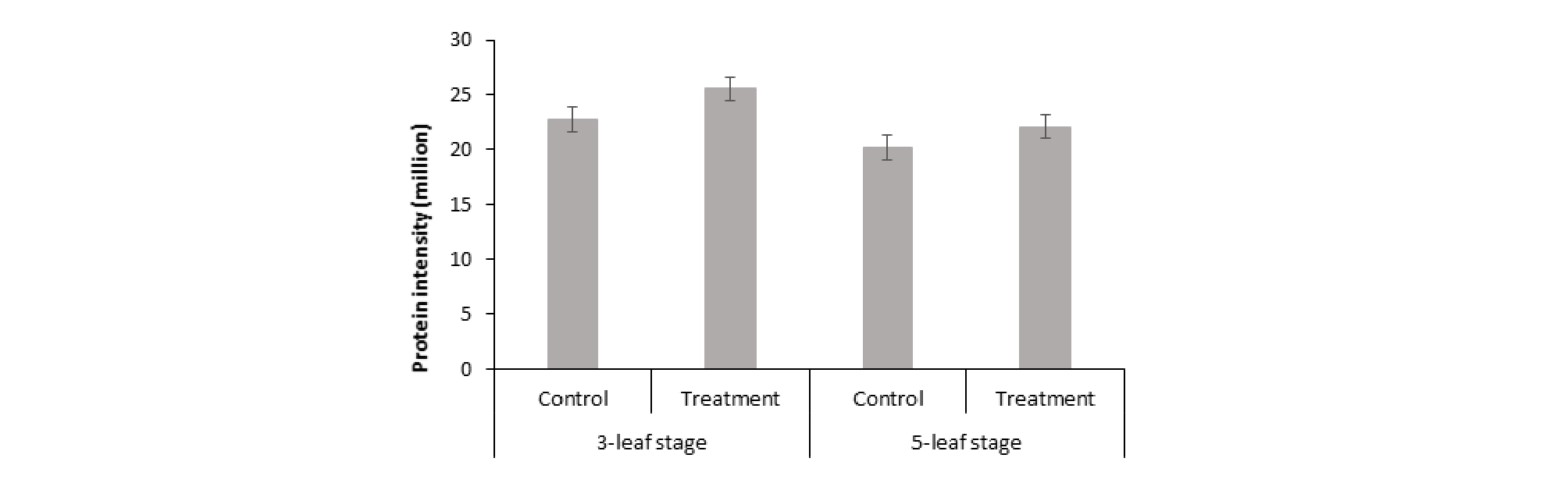
스팟 번호 773인 Superoxide dismutase [Mn] 3.4 단백질의 Gene은 SODA.3 이다. Superoxide dismutase (SOD)는 초과산화이온을 산소와 과산화수소로 바꿔 주는 불균등화 반응을 촉매 하는 효소로(2 superoxide + 2H+ = O2 + H2O2) 산소에 노출되는 거의 모든 세포에서 항산화방어기작을 하는 것으로 알려져 있다. Cofactor로 Mn2+ 이 있으며, 세포 내에서 활성 산소가 과량으로 생산되면 세포장해, 조직장해 등을 일으키는데, 이러한 원인 중 하나인 활성 산소(activated oxygens: AO)의 불균등화 반응을 촉매 하는 효소이다. 세포 내인성 보호 메커니즘은 superoxide dismutase와 같은 여러 효소를 포함하는데 이러한 효소들은 독성 산소 종을 효과적으로 제거할 수 있다(Ahmed et al., 2002). 미토콘드리아에 존재하면서 molecular function으로는 금속이온 결합, 과산화물 디스뮤타아제 활성이 있으며, biological process로는 ABA (abscisic acid)에 대한 반응, 삼투압 스트레스에 대한 반응이 있다.
Superoxide dismutase 활성의 증가는 과습 스트레스에 의한 산화 손상으로부터 식물의 보호에 필수적이다(Bowler et al., 1992). 이 단백질의 경우 무처리보다 과습 처리에서 더 많은 양이 발현된 것으로 보아(Fig. 11) SOD가 과습 스트레스에 대해서도 반응을 하는 것으로 보인다. 다시 말해 과습 스트레스를 받은 처리구의 식물체 내에서 활성 산소에 대한 항산화 기능을 하는 효소의 활성이 더 많아진다는 것을 의미한다. 따라서 과습 스트레스가 식물체 내 활성 산소 발생 증가의 원인 중 하나로 작용하는 것으로 사료된다.
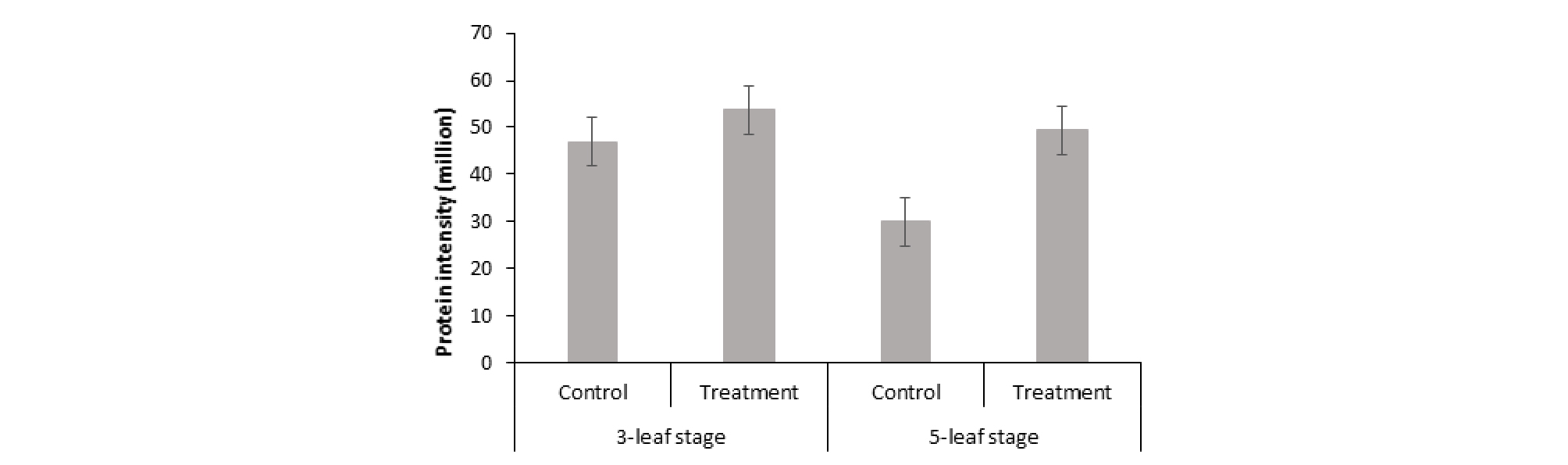
스팟 번호 764인 probable phospholipid hydroperoxide glutathione peroxidase는 CSA 유전자를 가지며, glutathione에 의해서 과산화수소, 지질 과산화물, 유기계 히드록시 과산화물의 감소를 촉매(2 glutathione + a hydroperoxy-fatty- acyl-[lipid] = glutathione disulfide + a hydroxy-fatty- acyl-[lipid] + H2O)함으로써 산화 손상으로부터 세포와 세포의 효소를 보호하는 역할을 하는 단백질로 알려져 있다(Sugimoto and Takeda, 2003). 세포 내 세포질 속에서 존재하며 molecular function으로는 glutathione peroxidase activity, phospholipid- hydroperoxide glutathione peroxidase activity가 있으며, biological process로는 산화 스트레스에 대한 반응이 있다.
이 단백질 역시 3엽기와 5엽기에서 모두 무처리보다 과습 처리에서 더 많이 발현되는 양상을 확인할 수 있었다(Fig. 12). 따라서 과습 스트레스로 인해서 세포에 손상이 유발됨을 알 수 있었고, 이에 반응하여 과습 처리구에서 해당 단백질의 발현양이 증가된 것으로 사료된다.
적요
밭작물 중의 하나인 수수의 재배기간 중 장기간 지속되는 토양의 과습 상태는 수수의 생장 저하를 야기하는 요소로서 작용한다. 수수 잎을 이용한 수수의 3엽기, 5엽기 과습 처리시의 단백질을 동정한 결과 74개의 단백질들을 동정하였다. LTQ-FI-ICR MS로 분석한 결과 carbohydrate metabolic process, metabolic process, cellular metabolic compound salvage와 관련된 단백질들이 약 50% 정도를 차지하며, 과습 스트레스를 받을 때 영향을 미치는 것으로 보였다. Carbohydrate metabolic process와 관련된 malate dehydrogenase 단백질과 glyceraldehyde-3- phosphate dehydrogenase 단백질은 과습 스트레스를 받았을 경우 3엽기와 5엽기 모두 단백질 발현양이 증가하였다. 이러한 단백질들은 과습 스트레스에 대한 antistress 기능을 하는 단백질로 알려져 있는데, 과습 스트레스에 반응하여 해당 단백질들의 발현양이 증가한 것으로 사료된다. 광합성과 관련된 oxygen- evolving enhancer protein 1 단백질은 대조구에서 보다 처리구에서 발현양이 다소 증가한 것을 볼 수 있었다. 또한, 3엽기에 비해서 5엽기에서 발현양이 증가한 것을 확인할 수 있었다. Response to oxidative stress와 관련된 probable phospholipid hydroperoxide glutathione peroxidase 단백질과 superoxide dismutase 단백질 모두 5엽기 처리구에서 가장 많은 발현량을 보였다. 이는 과습 스트레스에 의한 활성산소 발생이 5엽기 처리구에서 가장 많았고, 이에 따라 항산화방어기작을 하는 단백질의 발현이 증가한 것으로 여겨졌다.